B0-1 - Amminoacidi e Proteine
-
Upload
duchamps-lechataux -
Category
Documents
-
view
85 -
download
10
Transcript of B0-1 - Amminoacidi e Proteine
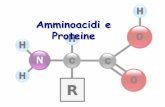
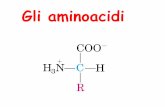
AMMINOACIDI
GRUPPO AMMINICO
GRUPPO CARBOSSILICO
Catena laterale
α
α-Amminoacidi
Gli amminoacidi sono composti che contengono sia il gruppo amminico che il gruppo carbossilico.
Amminoacidi
Nomenclatura
Nome IUPAC
Nome comune
Gli α-amminoacidi generalmente portano un nome comune e sono identificati da due tipi di abbreviazioni: a una e a tre lettere.
Gli atomi di carbonio della catena laterale sono designati con le lettere dell’alfabeto greco a partire dal carbonio α.
Esempio: LISINA
(Lys, K)
α
β
γ
δ
ε Nome IUPAC
Acido 2,6-diamminoesanoico
Amminoacidi
Gli amminoacidi più comuni presenti nelle proteine sono 20. Anche altri amminoacidi, ottenuti da modifiche post-traduzionali (esempi: idrossiprolina, idrossilisina,…) possono partecipare alla struttura delle proteine.
ESSENZIALI:
Valina, Leucina, Isoleucina, Triptofano, Lisina, Metionina, Fenilalanina, Treonina.
NON ESSENZIALI:
Glicina, Alanina, Prolina, Tirosina, Serina, Cisteina, Acido glutammico, Acido
aspartico, Istidina*, Arginina**.
*Istidina: essenziale per i bambini e non per gli adulti. **Arginina: sintetizzata ma in quantità non sufficienti per il fabbisogno metabolico .
Amminoacidi
Classificazione
In base alla struttura e alle proprietà chimiche delle catene laterali (R), gli amminoacidi possono essere suddivisi in:
Alifatici Aromatici ed eterociclici Neutri
Acidi Basici Polari
Non polari (idrofobici)
POLARITA’
La distribuzione non uniforme degli elettroni in un composto contenente legami covalenti viene misurata da una grandezza momento dipolare. Il momento dipolare (μ) è una grandezza vettoriale definita dalla seguente equazione:
μ =qr dove q è il valore della carica e r è il vettore distanza diretto dalla carica positiva alla carica negativa. Il momento dipolare è espresso in Debay (D). Si definiscono molecole polari le molecole che presentano momenti dipolari permanenti.
Amminoacidi
A
L
I
F
A
T
I
C
I
Glicina
(Gly, G)
Alanina
(Ala, A) Valina
(Val, V)
Isoleucina
(Ile, I)
AMMINOACIDI NEUTRI
(non polari)
Leucina
(Leu, L)
Metionina
(Met, M)
Prolina
(Pro, P)
A
R
O
M
A
T
I
C
I
Fenilalanina
(Phe, F)
Triptofano
(Trp, W)
Amminoacidi
A
L
I
F
A
T
I
C
I
AMMINOACIDI NEUTRI
(polari)
Serina
(Ser, S) Treonina
(Thr, T)
Asparagina
(Asn, N) Glutammina
(Gln, Q)
Cisteina
(Cys, C)
A
R
O
M
A
T
I
C
I
Tirosina
(Tyr, Y)
Amminoacidi
AMMINOACIDI ACIDI
AMMINOACIDI BASICI
Acido glutammico
(Glu, E)
Acido aspartico
(Asp, D)
Arginina
(Arg, R)
Lisina
(Lys, K)
Istidina
(His, H)
Amminoacidi
Proprietà acido-base Sostanze anfotere
SWITTERIONE
pI = PUNTO ISOELETTRICO
La carica complessiva è nulla
Carica netta +1
Predomina a pH << pI
Carica netta -1
Predomina a pH >> pI
Alanina
pKa1 = 2.35 pKa2 = 9.69
pI = 6.02
pI = pKa1 + pKa2
2
Amminoacidi
Proprietà acido-base
Acido aspartico
pKa1 = 1.88 pKa2 = 3.65 pKa3 = 9.60
Carica netta +1 Carica netta -1 Carica netta -2 Carica netta 0
pI = 2.76
pI = pKa1 + pKa2
2
= 1.88 + 3.65
2
= 2.76
Amminoacidi
Proprietà acido-base
AA R pKa1 pKa2 pKa3 pI
Ac. aspartico -CH2COOH 1.88 3.65 9.60 2.76
Ac. glutammico -CH2CH2COOH 2.16 4.32 9.67 3.24
Tirosina -CH2PhOH 2.20 9.11 10.07 5.65
Cisteina -CH2SH 1.71 8.18 10.28 5.02
Lisina -(CH2)4NH2 2.18 9.12 10.53 9.82
Arginina -(CH2)3NHC(NH2)=NH 2.17 9.04 12.48 10.76
Istidina -CH2-imidazolo 1.82 6.00 9.17 7.58
Amminoacidi basici
pI > pH neutro
Arginina Lisina Istidina meno
basico
più
basico
Amminoacidi acidi
pI < pH neutro
Carica netta negativa a pH neutro
Amminoacidi
Stereochimica
α
Con la sola eccezione della glicina,
il carbonio α degli amminoacidi è un
CENTRO STEREOGENICO.
MOLECOLA CHIRALE
molecola non sovrapponibile alla
propria immagine speculare.
180°
Amminoacidi
Stereochimica
Assegnazione della configurazione assoluta: sistema R, S
Regole di Cahn-Ingold-Prelog
• Assegnare la priorità ai quattro differenti gruppi
• Se il gruppo a priorità minore si trova sul cuneo tratteggiato si procede
tracciando una freccia dal gruppo a priorità maggiore a quello che segue in
ordine di priorità. Senso orario: configurazione R. Senso antiorario:
configurazione S.
• Se il gruppo a priorità minore non si trova sul cuneo tratteggiato
scambiare tra loro due gruppi in modo tale da portare il gruppo con priorità
4 in tale posizione. Si ottiene così l’enantiomero della molecola di partenza
la cui configurazione assoluta può essere assegnata come sopra. Se si ottiene
configurazione R allora la molecola di partenza ha configurazione S e
viceversa.
1
2
3
Configurazione S
Il carbonio α degli amminoacidi in natura ha configurazione S con l’eccezione della cisteina
Amminoacidi
Stereochimica
Gli amminoacidi in natura hanno configurazione L.
(S)-alanina
1
2
3
(S)-L-alanina
Proiezioni di Fischer
Proiezioni di Fischer
Notazione D, L
Formule prospettiche
Amminoacidi
Attività ottica
Un composto capace di ruotare il piano della luce polarizzata di definisce
OTTICAMENTE ATTIVO. Le molecole chirali sono otticamente attive.
Rotazione del piano della luce polarizzata
In senso
DESTROGIRO
(+) o d
In senso
LEVOGIRO
(-) o l
(S)-(+)-alanina (R)-(-)-alanina
L-alanina D-alanina
Amminoacidi
Conformazioni
Proiezioni di Newman
Analisi conformazionale
Isoleucina
Conformeri eclissati
Conformeri sfalsati
Gauche + Gauche - Anti
Angolo diedro
Più stabile
PEPTIDI E PROTEINE
Legame peptidico
Legame covalente che si forma per
reazione tra i gruppi α-carbossilico di un
amminoacido e α-amminico di un secondo
amminoacido.
La reazione avviene mediante eliminazione
di una molecola di acqua e il legame che si
forma è un legame ammidico.
Legame peptidico
Estremità
N-terminale Estremità
C-terminale
Peptidi e proteine Legame peptidico
• Il legame peptidico può essere descritto come ibrido di due forme tautomeriche
limiti (il legame ha caratteristiche intermedie tra legame semplice e doppio) attraverso
la risonanza.
Forma tautomerica aci-ammidica
risonanza risonanza
Struttura mesomerica
Peptidi e proteine Legame peptidico
• Non è possibile la rotazione libera intorno al legame peptidico a causa del
parziale carattere di doppio legame. La configurazione trans è più stabile.
Possono invece ruotare liberamente il legame C-C di un amminoacido il quale forma
un angolo di rotazione detto “psi” e il legame N-C di un altro amminoacido con
angolo di rotazione “phi”.
La rotazione dipende dall’ingombro sterico delle catene laterali
Peptidi e proteine Legame peptidico
Configurazioni cis e trans
Peptidi e proteine
Generalmente la configurazione
trans è più stabile
•Per ragioni di ingombro sterico la
sequenza aa-Pro si ritrova in
configurazione cis.
Peptidi e proteine
Legame peptidico
• Gli atomi di carbonio e di azoto
coinvolti nel legame peptidico e gli
atomi ad essi legati giacciono su un
piano. La planarità influenza il modo
in cui le catene laterali si ripiegano
nella struttura tridimensionale delle
proteine.
• La parziale separazione di carica nel legame
peptidico genera un dipolo elettrico.
Peptidi e proteine
Nomenclatura
Estremità
N-terminale
Estremità
C-terminale
Ala-Cys-Val (oppure A-C-V)
Alanilcisteilvalina
Scheletro peptidico
o backbone
Proprietà
Le proprietà caratteristiche di un peptide o di
una proteina dipendono dalla struttura e dal
numero di catene laterali
Sulla base del numero di residui amminoacidici si distinguono:.
Dipeptide, tripeptide
pentapeptide …….
Oligopeptide Polipeptide
Proteina
Proteine
Struttura delle proteine
STRUTTURA PRIMARIA
Sequenza di amminoacidi che costituisce la proteina.
…-Gly-Val-Leu-His-Arg-Thr-…
STRUTTURA SECONDARIA
Disposizione nello spazio dello scheletro peptidico
α-elica, foglietto β, …
STRUTTURA TERZIARIA
Modo in cui la catena o le catene polipeptidiche si ripiegano su se stesse in seguito ad
interazioni intramolecolari dando strutture più complesse come le strutture sferoidali
delle proteine globulari.
STRUTTURA QUATERNARIA
Aggregazione di due o più catene polipeptidiche (subunità) tramite interazioni per lo
più non covalenti.
Proteine
STRUTTURA SECONDARIA
STRUTTURA PRIMARIA
…-Gly-Val-Leu-His-Arg-Thr-… Ordine con cui i diversi amminoacidi si
susseguono nella catena polipeptidica.
Disposizione nello spazio dello scheletro peptidico
Resa stabile da legami idrogeno che coinvolgono gli atomi dello scheletro
peptidico.
La conformazione dello scheletro peptidico generalmente definita dagli angoli
di rotazione “phi” e “psi” (-180° a +180°) per ogni residuo amminoacidico.
Resa stabile da legami covalenti.
I tipi di strutture secondarie che si
ripetono più frequentemente nelle
proteine sono:
α-elica
foglietto β
Proteine
α-elica
STRUTTURA SECONDARIA
• Coinvolge una singola catena polipeptidica
• È stabilizzata da legami idrogeno paralleli
all’asse dell’elica
• Il legame idrogeno coinvolge il gruppo
C=O di un amminoacido n e il gruppo NH
dell’amminoacido n+4.
• Gli atomi coinvolti nel legame idrogeno
assumono una disposizione lineare.
• Le catene laterali si dispongono verso
l’esterno dell’elica.
• Un giro d’elica è costituito da 3.6 residui e
il passo dell’elica è di 5.4Å.
Proteine STRUTTURA SECONDARIA
foglietto β
(β sheet)
Foglietto β antiparallelo
• Coinvolge una o più catene polipeptidiche
• L’unità costituente i foglietti β è il
filamento β.
• Il legame idrogeno coinvolge il gruppo
C=O di un amminoacido e il gruppo NH
dell’amminoacido del filamento β adiacente.
• I legami idrogeno danno origine ad una
struttura ripetitiva a zig zag.
• Si distinguono in foglietti β paralleli,
antiparalleli e misti.
Foglietto β parallelo
Proteine
L’inversione della direzione della catena polipeptidica è possibile in presenza di
un elemento strutturale definito reverse turn (o ripiegamento inverso).
β-turn
Tipo I
Il legame idrogeno si forma tra il gruppo C=O del residuo n e il gruppo NH del residuo
n+3. Due residui non sono interessati da legame idrogeno.
Tipo II
La differenza principale tra i tipi I e II
consiste nell’orientazione relativa del legame
peptidico tra gli amminoacidi n+1 e n+2.
Tipo III
γ-turn Ω-loop
Proteine Reverse turn
Infine, i segmenti di catena polipeptidica che non assumono una struttura secondaria definita
sono detti ‘random coil’. Sono strutture irregolari, con angoli diedri di ampiezza variabile .
‘Random coil’
Il legame idrogeno interessa il gruppo
C=O del residuo n e il gruppo NH del
residuo n+2. Soltanto un amminoacido
non è interessato dal legame idrogeno.
Strutture secondarie non regolari con
numero di legami idrogeno e ampiezze degli
angoli diedri variabili. Presentano struttura
compatta e non disordinata, con le catene
laterali rivolte verso l’interno della struttura
rendendola stabile. Sono frequenti nelle
proteine globulari. Spesso hanno un ruolo
nel riconoscimento intermolecolare.
Angoli inusuali per n+1
Proteine
STRUTTURA TERZIARIA
La struttura terziaria di una proteina è la disposizione tridimensionale di tutti gli atomi
della molecola. Ha origine dal ripiegamento delle strutture secondarie.
Interazioni : -Legami idrogeno
-Interazioni idrofobiche
-Interazioni elettrostatiche
-Interazioni di Van der Waals
-Ponti disolfuro
Interazioni deboli
Legame covalente
La struttura terziaria è resa stabile da interazioni tra le catene laterali degli
amminoacidi. Si tratta principalmente di interazioni deboli con l’eccezione del
legame disolfuro tra residui di Cys che è un tipo di legame covalente.
Elementi di
struttura secondaria
Strutture
supersecondarie
o motivi
Domini
STRUTTURA
TERZIARIA
Proteine
STRUTTURA QUATERNARIA
Aggregazione di due o più catene polipeptidiche (subunità) tramite interazioni per lo
più non covalenti. Le interazioni coinvolte sono analoghe a quelle che si osservano
nelle strutture terziarie: interazioni elettrostatiche, interazioni idrofobiche, legami
idrogeno e ponti disolfuro.
In funzione del numero di subunità si distinguono in proteine oligomeriche
(dimeri, trimeri,…) e proteine multimeriche. Se le subunità sono uguali si
definiscono proteine omomultimeriche altrimenti proteine eteromultimeriche.
VEGF
omodimero
Proteine
Classificazione delle proteine
basata sulla forma
Proteine GLOBULARI Proteine FIBROSE
• Forma sferica o globulare
• AA idrofobici all’interno e idrofilici
all’esterno
• Solubili in acqua (fluidi biologici)
• Costituite da diverse strutture secondarie
• Funzioni biologiche: funzione dinamica
(catalisi, trasporto …)
• Più tipi
• Esempi: emoglobina e mioglobina
• Forma lineare, semplice e regolare
• Tendenza ad aggregarsi dando strutture
macromolecolari (fibre, foglietti …)
• Insolubili in acqua
• Generalmente costituite da un unico tipo
di struttura secondaria
• Funzioni biologiche: funzione strutturale e
protettiva
• Massa totale maggiore
• Esempi: cheratina e collagene
Proteine di MEMBRANA
Proteine:
legami e interazioni
Proteine
Legami covalenti
Legame disolfuro
Altre interazioni
Legami idrogeno
Interazioni elettrostatiche (legami ionici e interazioni dipolo-dipolo)
Interazioni di Van der Waals
Interazioni idrofobiche
Legame peptidico (legame ammidico)
LEGAME COVALENTE
Legame chimico formato da due atomi che condividono una
o più coppie di elettroni.
Legame covalente polare: Gli elettroni di legame non sono
equamente condivisi. La differenza di elettronegatività tra gli
atomi è compresa tra 0.5 e 1.9.
Legame covalente non polare: Gli elettroni di legame
sono equamente condivisi. La differenza di elettronegatività
tra gli atomi è inferiore a 0.5.
Proteine
LEGAME DISOLFURO
Il legame disolfuro si forma per reazione di
ossidazione di gruppi tiolici.
Nelle proteine tale legame si forma tra residui di
cisteina per formare una molecola definita cistina.
Importante per la stabilizzazione intra ed inter-
catene di proteine.
Proteine
LEGAME IDROGENO
Il legame idrogeno è un’interazione dipolo-dipolo tra un atomo di idrogeno legato
covalentemente ad un atomo elettronegativo (donatore di legame a idrogeno) e un
altro atomo elettronegativo che presenta una coppia di elettroni non condivisa
(accettore di legame a idrogeno). E’ un’interazione debole.
Donatore di
legame idrogeno
Legami idrogeno che interessano una singola molecola sono definiti intramolecolari,
se tra molecole diverse intermolecolari.
Accettore di
legame idrogeno
Nelle proteine …
La lunghezza del legame è quasi doppia a quella dei legami covalenti.
Nelle proteine i legami idrogeno coinvolgono frequentemente i gruppi C=O
(accettore) e NH (donatore) dello scheletro peptidico.
.
Proteine
Potenziali gruppi donatori di legami idrogeno:
Es. -OH (Ser, Tyr, Thr) –SH (Cys) - NHn
Principali gruppi coinvolti in legami idrogeno
I gruppi accettori di legame idrogeno contengono atomi elettronegativi (come
ossigeno e azoto) con un coppia di elettroni non condivisa.
Es. -COO-
.
Proteine
INTERAZIONI ELETTROSTATICHE
Le interazioni elettrostatiche sono interazioni non covalenti che coinvolgono gruppi
carichi.
Le forze di attrazione elettrostatica tra cariche nette (ioni) determinano la formazione
di sali (legami ionici). Esempio: interazione tra atomi di azoto con carica positiva (Lys,
His, Arg) e ioni carbossilato (Glu, Asp).
Le interazioni dipolo-dipolo sono generate da forze di attrazione tra l’estremità
positiva di un dipolo e quella negativa di un altro dipolo. Sono interazioni deboli che
coinvolgono molecole con carica parziale.
Le forze di repulsione allontanano cariche nette di segno uguale.
Le interazioni idrofobiche sono interazioni deboli che coinvolgono residui
idrofobici (nuclei aromatici e catene alchiliche) che tendono ad interagire tra loro per
effetto della stessa natura non polare.
Partecipano alla conformazione e stabilità delle proteine.
Proteine
INTERAZIONI IDROFOBICHE
INTERAZIONI DI VAN DER WAALS
Le interazioni di Van der Waals si originano per azione di deboli forze attrattive
(forze di Van der Waals o forze di dispersione) tra dipoli istantanei di atomi o
molecole vicini. Possono essere definite interazioni dipolo – dipolo indotto.
Possono essere coinvolti in interazioni di Van der Waals e interazioni
idrofobiche gli amminoacidi neutri non polari.
Peptidi
Ormone prodotto dall’ipotalamo e secreto dalla neuroipofisi.
Peptide costituito da 9 amminoacidi.
Ossitocina
Stimola la contrazione della
muscolatura uterina e la
lattazione per azione sulla
ghiandola mammaria.
Sequenza primaria:
Ponte disolfuro tra i residui di cisteina 1 e 6
genera un ciclo a 20 atomi
Tirosina
Isoleucina
Asparagina
L’ossitocina presenta
scarsa stabilità metabolica