PROTEINE E PROTEOMICA CLINICA - medinfo.dist.unige.itInfo_BT\P_02_Allegati.pdf · La proteomica è...
Transcript of PROTEINE E PROTEOMICA CLINICA - medinfo.dist.unige.itInfo_BT\P_02_Allegati.pdf · La proteomica è...

Gestione dei dati in proteomica
1
Gestione dei dati in proteomica
PROTEINE E PROTEOMICA CLINICA
1.1 Le proteine: natura e funzioni
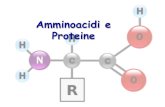
Le proteine sono i prodotti della policondensazione di numerose molecole α-amminoacidi,
legate le une alle altre mediante legami ammidici (legame peptidico) tra il gruppo carbossilico
di una molecola ed il gruppo amminico della molecola successiva, a costituire una catena
polipeptidica (fig. 1).
Figura 1: (1) amminoacidi in forma neutrale; (2) nella forma in cui esistono fisiologicamente;
(3) legati insieme a formare un dipeptidi.
A seconda del numero di molecole di amminoacido si può avere formazione di dimeri,
trimeri, tetrameri e così via fino ai polimeri costituiti da molte migliaia di molecole di
monomeri. La distinzione fra proteine e polipeptidi è alquanto convenzionale ed incerta:
generalmente si considerano polipeptidi i polimeri aventi peso molecolare fino a circa
5000/6000 dalton, e proteine quelli con peso molecolare maggiore, fino ad un valore di
parecchi milioni di dalton [1]. Gli amminoacidi conosciuti in natura sono solo venti, ma dalla
loro combinazione si generano migliaia e migliaia di peptidi, polipeptidi e proteine.
Mentre ci sono solo venti differenti tipi di amminoacidi che danno origine a tutte le proteine,
a volte servono centinaia di essi per fare una singola proteina. Aggiunta a questa complessità,

Gestione dei dati in proteomica
2
tipicamente le proteine non rimangono sotto forma di lunghe catene. Non appena la catena di
amminoacidi è costruita, la catena si ripiega e si aggroviglia in una forma particolare e più
compatta che le permette di effettuare specifiche e necessarie funzioni nel corpo umano. Le
proteine si ripiegano perché i differenti amminoacidi preferiscono attaccarsi l'un l'altro
secondo alcune regole di chimica e termodinamica [2].
Le proteine sono essenziali per gli essere viventi; ogni attività del corpo umano coinvolge le
proteine e, di conseguenza, la loro conoscenza risulta fondamentale per una buona
comprensione della fisiologia e dei processi dell’organismo umano. Nella stesura di questo
lavoro viene focalizzata l’attenzione sulle proteine plasmatiche presenti nel sangue umano.
Esse sono le responsabili dell’alta viscosità relativa del plasma pari a 1,9-2,6 (l’acqua è 1),
essendo presenti in quantità pari a 65-80 g/l. I pesi molecolari dei ciascuna proteina
plasmatica oscillano tra 44.000 e 1.300.000 dalton (particelle di tale ordine appartengono alla
classe dei colloidi).
Le proteine plasmatiche assolvono a molte funzioni, di cui elenco le principali [3]:
• Nutritiva: nei circa tre litri di plasma dell’individuo adulto si trovano disciolti 200g di
proteine; questa quantità costituisce una riserva proteica rapidamente disponibile. Le proteine,
una volta scisse negli amminoacidi, sono quindi una riserva per l’intero organismo;
• Di trasporto: grazie al legame dei loro gruppi liofili a sostanze lipidiche non solubili in
acqua, le molecole proteiche plasmatiche funzionano come mezzo solvente;
• Genesi della pressione oncotica: sono molto importanti per il mantenimento appunto della
pressione oncotica (PCO): da notare il fatto che, essendo l’albumina la componente maggiore
delle proteine plasmatiche, le variazioni della sua concentrazione producono effetti
particolarmente rilevanti sulla pressione oncotica;
• Tampone: essendo le proteine anfoliti, quelle plasmatiche funzionano come tampone
contribuendo al mantenimento di un valore costante del pH;
• Antiemorragica: la coagulabilità del sangue dipende dalla presenza della proteina plasmatica
fibrinogeno;
• Di difesa: alcune proteine servono al riconoscimento specifico e non, e quindi alla
distruzione di corpi estranei e agenti patogeni.
Da questo rapido excursus si evince chiaramente come la quantità, la struttura ed infine la
funzione delle diverse proteine contenute nell’organismo umano siano aspetti di primaria
importanza, legati alle basilari funzioni che esse assolvono. Nel corso degli ultimi decenni le

Gestione dei dati in proteomica
3
innovazioni in campo tecnologico ed informatico hanno permesse di compiere passi in avanti
notevoli nello studio identificativo e sulla natura delle relazioni esistenti intra ed extra le catene
proteiche.
1.2 Proteoma e Proteomica
Il sequenziamento dell’intero genoma di alcuni organismi, principalmente quello umano, e lo
sviluppo e i progressi nei metodi e nelle tecnologie di analisi, hanno aperto nuovi scenari di
indagine conferendo alle proteine un ruolo sempre più importante che sta suscitando un
interesse sempre maggiore presso la comunità scientifica internazionale [4].
Si definisce “genoma umano” la collezione di tutti i geni, il cui numero esatto è ancora
dibattuto, ma è sicuramente superiore alle 30000 unità. Ogni gene, che è una sezione di una
lunga catena nota come DNA, detta come costruire la catena di amminoacidi per una diversa
proteina. Questo significa che è possibile conoscere la sequenza di amminoacidi in tutte le
proteine umane. Questa relazione causale evidenzia quindi lo stretto legame fra il genoma di
tutti gli esseri viventi e le proteine di cui sono costituiti.
L’attenzione al mondo delle proteine ha reso necessaria la nascita di un’ontologia che
permettesse di riferirsi al nuovo campo di ricerca; nel 1994 Mark Wilkins coniò dunque il
termine proteoma, termine che descrive l’insieme delle proteine di un organismo o di un
sistema biologico, ovvero le proteine prodotte dal genoma. Si può considerare il proteoma
completo di un organismo, che può essere immaginato come l'insieme globale delle proteine
di tutti i proteomi cellulari. Questo è, quindi, per analogia, l'equivalente proteico del genoma.
Il proteoma è più grande del genoma, specialmente negli eucarioti, dal momento che,
numericamente, ci sono più proteine che geni: ciò è dovuto all'accoppiamento dei geni ed alle
modificazioni post-traslazionali come la glicosilazione o la fosforilazione Il proteoma mostra
almeno due livelli di complessità che mancano al genoma. Mentre il genoma è definito da una
sequenza di nucleotidi, il proteoma non si limita alla somma delle sequenze di proteine
presenti. Infine, la conoscenza del proteoma richiede di conoscere, oltre alle strutture delle
proteine del proteoma, anche le interazioni funzionali tra le proteine stesse. Di particolare
interesse è inoltre il fatto che, a differenza del "genoma", che può essere considerato
virtualmente statico, il "proteoma" cambia continuamente. Alcune patologie, come ad esempio

Gestione dei dati in proteomica
4
i tumori, provocano drastici cambiamenti nella composizione delle proteine normalmente
espresse e,come effetto, cambiano anche le interazioni fra proteina e proteina [5].
Lo studio del proteoma si chiama proteomica, per analogia, anche in questo caso, al termine
genomica.
La proteomica è una disciplina scientifica che mira ad identificare le proteine ed ad associarle
con uno stato fisiologico in base all'alterazione del livello di espressione fra controllo e
trattato. Permette di correlare il livello di proteine prodotte da una cellula o tessuto e l'inizio o
la progressione di uno stato di stress. La proteina "segnale" identificata con un approccio
proteomico ha un ampio spettro di potenziali applicazioni. Può essere usata per lo sviluppo di
nuovi "biomarker" o per lo studio della funzione di un gene. Le proteine possono essere
utilizzate per osservare gli effetti di specifici trattamenti o inquinanti ambientali.
L'abbondanza di informazioni fornite da una ricerca proteomica sono complementari con le
informazioni genetiche generate da ricerche genomiche. La proteomica, infatti, sarà cruciale
per lo sviluppo della genomica funzionale. La combinazione di proteomica e genomica sta
giocando e giocherà in futuro un ruolo fondamentale nella ricerca biomedica e avrà un impatto
significativo sullo sviluppo dei sistemi diagnostici.
La proteomica è una scienza, come si può intendere facilmente, giovanissima, finora vissuta
quasi esclusivamente nei laboratori, ma che adesso sta incominciando ad essere applicata
sperimentalmente sui pazienti diventando così proteomica clinica.
1.3 Ambiti di sviluppo della proteomica Le principali branche della proteomica “di ricerca” e, quindi, non direttamente clinica, e le
principali tecniche utilizzate sono le seguenti [6]:
1. Separazione di proteine. Tutte le tecnologie della proteomica risiedono sulla capacità di
separare da una miscela complessa singole proteine in modo che possano essere processate
con ulteriori tecniche.
2. Identificazione di proteine. Metodi comuni "low-throughput" includono il sequenziamento
mediante degradazione Edman. Metodi "high-throughput" sono basati su spettrometria di
massa, peptide mass fingerprinting o sequenziamento ‘De novo repeat detection’.

Gestione dei dati in proteomica
5
Possono anche essere usati saggi basati su anticorpi, ma sono diretti unicamente verso un
singolo epitopo.
3. Quantificazione di proteine. Esistono metodi basati sul gel con marcatura fluorescente
(Cy3, Cy5) (gel elettroforesi differenziale) e metodi "gel-free", che includono metodi di
"tagging" o di modificazione chimica, come "isotope-coded affinity tags" (ICATs) o
"combined fractional diagonal chromatography" (COFRADIC).
4. Analisi di sequenza di proteine. Questa è una branca prettamente bioinformatica, rivolta
alla ricerca nelle banche dati, per l'identificazione della proteina o peptide. Da questo tipo di
analisi di sequenza possono essere tratte anche informazioni di carattere funzionale ed
evolutivo (attraverso il multiallineamento delle proteine).
5. Proteomica strutturale. Questa parte delle proteomica si occupa dello studio
tridimensionale delle proteine usando metodi di cristallografia a raggi X e spettroscopia
NMR.
6. Studio delle interazioni fra proteine. Studio delle interazioni fra proteine a livello atomico,
molecolare e cellulare.
7. Modificazioni post-traduzionali delle proteine. Questa branca della proteomica si occupa
dello studio delle modificazioni che le proteine subiscono dopo essere state tradotte. Allo
scopo sono stati sviluppati metodi adeguati per studiare la fosforilazione
("fosfoproteomica") e la glicosilazione ("glicoproteomica").
8. Proteomica cellulare. Nuova branca della proteomica il cui scopo principale è quello di
mappare la localizzazione delle proteine e delle interazioni fra proteine nelle cellule durante
particolari "eventi-chiave" della vita cellulare. Le tecniche usate fanno capo alla "X-Ray
Tomography" e alla microscopia ottica a fluorescenza.
1.4 La gestione dei dati in proteomica Lo studio proteomico richiede il continuo sviluppo di metodi per il miglioramento delle
capacità separative, della sensibilità e delle possibilità di interpretazione dei dati correlati ai
segnali biologici; inoltre a causa della complessità delle relazioni e dei comportamenti che le
proteine instaurano con i diversi tessuti ed organi coinvolti e, infine, per l’importanza nella
attività che vanno a svolgere, i più grandi centri di ricerca scientifica internazionali
(accademici e non) si sono dotati di potenti banche di dati per raccogliere, catalogare e gestire
il maggior numero di informazioni possibili.

Gestione dei dati in proteomica
6
Ogni database ha, però, delle peculiarità e dei filoni preferenziali di ricerca diversi, per cui
soltanto con un attento e minuzioso lavoro di collazione tra le diverse informazioni contenute
in ciascuno, sarebbe possibile avere una conoscenza totale dello stato dell’arte della proteina e
di tutti i suoi derivati.
Un potente contributo alla diffusione della conoscenza nel campo della proteomica, sia dal
punto di vista delle informazioni scambiate, sia da quello della condivisione delle risorse è
stato fornito da metà degli anni ’90 in poi da Internet. Il mezzo telematico ha, infatti,
permesso, ai grandi istituti di ricerca e alle prestigiose università internazionali di “mettere in
rete” i loro lavori, di immagazzinare nuove conoscenze, di progredire nella ricerca in modo
pià rapido e più approfondito.
Allo stesso tempo, però, Internet ha portato un problema legato alla enormità delle
informazioni ora disponibili ai singoli utenti: districarsi all’interno di questa enorme mole di
dati composta da sequenze, codici identificatori, databases strutturati in modo differente può
portare ad un rifiuto ed ad un passo indietro nella ricerca.
In questo lavoro si è compiuta un’attenta ricerca sul web di tutte le banche di dati che si
“interessano” di proteine e di proteomica che fanno capo ai più importanti centri
internazionali: ne sono stati individuati una trentina, ognuno con determinate caratteristiche e
filoni di ricerca che ora vengono presentati in ordine di importanza. L’importanza, variabile
apparentemente soggettiva, è relativa alla qualità ed alla quantità delle informazioni presenti
nel database ed anche alla frequenza con cui i codici identificatori delle proteine vengono
“trovati” nelle ricerche sul web.
UniProt The universal protein resource (http://www.ebi.uniprot.org)
È il più grande catalogo al mondo di informazioni sul mondo delle proteine. È il “deposito
centrale” della sequenza e della funzione delle proteine generate unendo le informazioni
contenute in Swiss-Prot, in TrEMBL e in PIR (vedi successivamente). UniProt è composto da
tre componenti, ciascuno ottimizzato per un uso differente (UniProtKB, UniRef, UniParc, vedi
successivamente).

Gestione dei dati in proteomica
7
UniProtKB UniProt Knowledgebase (http://www.ebi.uniprot.org) È il punto di accesso centrale per informazioni accurate e precise sulle proteine, che
includono funzione, classificazione e cross-references. E’ una sotto sezione dell’UniProt
generale descritto precedentemente.
Vega (http://vega.sanger.ac.uk)
The Vertebrate Genome Annotation (VEGA) è il “deposito centrale” per l’alta qualità, gli
aggiornamenti frequenti e l’annotazione manuale della sequenza del genoma umano; i
particolari dei progetti per ogni specie sono disponibili attraverso le diverse homepage per
l'essere umano, il topo, il maiale ed il cane.
NCBI RefSeq (http://www.ncbi.nlm.nih.gov/RefSeq)
La collezione RefSeq mira a fornire un insieme integrato, completo, non ridondante delle
sequenze, includendo il DNA genomico, l’RNA trascritto e i derivati proteici per i più
importanti organismi di ricerca. I campioni di RefSeq servono come base per studi medici,
funzionali e di diversità; forniscono un riferimento stabile per l'identificazione e descrizione
del gene, analisi di mutazione, studi di espressione, scoperta di polimorfismo ed analisi
comparative. RefSeqs è usato come reagente per l'annotazione funzionale di un certo genoma
che ordina i progetti in serie, compreso quelli dell'essere umano e del topo.
Ensembl (http://www.ensembl.org)
È un progetto che coinvolge EMBL, EBI e il Sanger Istitute per sviluppare un sistema di
software che produca ed effettui l'annotazione automatica sui genomi eucariotici selezionati.

Gestione dei dati in proteomica
8
H-InvDB (http://www.h-invitational.jp/)
È un database integrato di geni umani; si appoggia sulla conoscenza delle banche di dati del
Giappone e compie ricerche soprattutto sull’RNA trascritto.
UniParc (http://www.ebi.uniprot.org) L'archivio di UniProt-UniParc è un deposito completo, che mostra la storia di tutte le sequenze
della proteina.
HGNC (http://www.gene.ucl.ac.uk)
Si propone di dare nomi unici ed espressivi al gene umano.
Entrez Gene (http://www.ncbi.nlm.nih.gov)
Entrez Gene è una base consultabile di dati dei geni, dei genomi di RefSeq e definito dalla
sequenza e situato nella viewer del programma di NCBI.
UniGene (http://www.ncbi.nlm.nih.gov)
UniGene è una vista organizzata del Transcriptome. Ogni record di UniGene è un insieme
delle sequenze della trascrizione che sembrano venire dallo stesso luogo della trascrizione
(gene o pseudogene espresso), insieme alle informazioni sulle somiglianze della proteina,
sull'espressione del gene, sui reagenti del clone del cDNA e sulla posizione genomica.

Gestione dei dati in proteomica
9
CCDS (http://www.ncbi.nlm.nih.gov/CCDS)
Il progetto dei CD di consenso (CCDS) è uno sforzo di collaborazione per identificare un
insieme di nucleo delle regioni di codificazione della proteina del topo e dell'essere umano che
sono annotate di frequente ed ad un livello di alta qualità. L'obiettivo di lunga durata è di
sostenere la convergenza verso un insieme standard delle annotazioni del gene.
Trome (http://ch.embnet.org/software/fetch.html)
Trome è una nuova base di dati che usa gli allineamenti dei dati di EST (expressed sequenze
tag) a HTG (high-throughput menome) e dei genomi completi per generare le trascrizioni e le
sequenze di codificazione virtuali. Questa nuova base di dati è di una qualità maggiore e,
poiché contiene le informazioni in una disposizione molto più densa, essa è di gran lunga
molto inferiore, in termini dimensionali, rispetto alle due basi di dati più piccole.
UtrDB (http://www2.ba.itb.cnr.it/UTRSite)
UTRSite è una collezione di modelli di sequenza funzionale (legati in
particolare all’RNA messaggero) situati nelle posizioni 5 ' o 3 '. Le proteine che si legano al 3'
o al 5' UTR possono danneggiare la traduzione interferendo con l'abilità dei ribosomi di
legarsi all'mRNA.

Gestione dei dati in proteomica
10
InterPro (http://www.ebi.ac.uk/interpro)
InterPro è una base di dati delle famiglie delle proteine, dei domini e delle
posizioni funzionali in cui le caratteristiche identificabili trovate nelle proteine conosciute
possono essere applicate alle sequenze sconosciute della proteina.
PFam (http://www.sanger.ac.uk/Software/Pfam/iPfam)
iPfam è una risorsa che descrive le interazioni di dominio-dominio che sono osservate nei
record di PDB. I domini sono definiti da Pfam. Quando due o i
più domini si presentano in una singola struttura, essi sono
analizzati per vedere se formano un'interazione. Se invece sono
abbastanza vicini formare un'interazione, si calcolano i legami che
l’interazioneproduce.
SMART (http://smart.embl-heidelberg.de/)
SMART sta per Simple Modular Architecture Research Tool; si può usare SMART in due
modi differenti: normale o genomic. La differenza principale è nella base di dati di fondo della
proteina usata. In SMART normale, la base di dati contiene lo Swiss-Prot, PS-TrEMBL e
proteomi stabili di Ensembl.

Gestione dei dati in proteomica
11
PROSITE (http://www.expasy.org/prosite)
PROSITE è una base di dati delle famiglie e dei domini della proteina. E’ formata dai modelli
e dai profili biologicamente significativi che contribuiscono ad identificare attendibilmente a
quale famiglia conosciuta della proteina (se esiste) una nuova sequenza appartiene.
CleanEx (http://www.cleanex.isb-sib.ch/)
CleanEx è una base di dati che fornisce l'accesso ai dati pubblici di espressione del gene
attraverso i simboli approvati unici del gene e che rappresenta i dati eterogenei di espressione
redatti dalle tecnologie differenti facilitando i confronti tra dataset di cross.references diversi.
PHANTER (http://www.pantherdb.org/)
Il sistema di classificazione PHANTER (Protein ANalysis THrough Evolutionary
Relationships) è una risorsa unica che classifica i geni per le loro funzioni, usando la prova
sperimentale scientifica pubblicata ed i rapporti evolutivi per predire la funzione anche in
assenza di prova sperimentale diretta. Per un numero crescente di proteine, le interazioni

Gestione dei dati in proteomica
12
biochimiche dettagliate nelle vie canoniche sono bloccate e possono essere osservate con
diverse interazioni.
PRINTS (http://umber.sbs.man.ac.uk/dbbrowser/PRINTS/)
PRINTS è un compendio di impronte digitali della proteina. Un'impronta digitale è un gruppo
dei motivi conservati usati per caratterizzare una famiglia della proteina; I motivi non
coincidono solitamente, ma sono separati lungo una sequenza, benchè possano essere attigui
nello spazio tridimensionale.
PIR (http://pir.georgetown.edu/)
Il Protein Information Resource si trova presso l’Università di GeorgeTown a Washington
DC ed è un centro di ricerca bioinformatico avanzato particolarmente nello studio della
proteomica e della genomica.
EPD (http://www.epd.isb-sib.ch/index.html)

Gestione dei dati in proteomica
13
L’Eukaryotic Promoter Database è una collezione non-ridondante annotata di promotor
eucariotici del POL II (che trascrive RNA per proteine), per cui il punto di inizio della
trascrizione è stato determinato sperimentalmente. L'accesso alle sequenze del promotor è
fornito dagli indicatori alle posizioni nelle entrate di sequenza del nucleotide. La parte di
annotazione di un'entrata include la descrizione dei dati di tracciato di luogo di inizio, dei
riferimenti ad altre basi di dati e dei riferimenti bibliografici. EPD è strutturato in modo da
facilitare l'estrazione dinamica dei sottoinsiemi biologicamente espressivi del promotor per
l’analisi comparativa di sequenza.
TIGRFAMs (http://www.tigr.org/TIGRFAMs/)
TIGRFAMs sono famiglie di proteine basate sugli Hidden Markv Models.
Superfamily (http://supfam.org/SUPERFAMILY/)
Lo scopo di questo server è fornire assegnazioni funzionali e strutturale alle sequenze della
proteina al livello del superfamily. Una superfamily contiene tutte le proteine per cui si ha
prova strutturale di un antenato evolutivo comune.
Transfac (http://www.genome.ad.jp/dbget-bin/www_bfind?transfac)

Gestione dei dati in proteomica
14
È una banca di dati che fa riferimento al centro bioinformatico dell’Università di Kyoto.
ProDom (http://prodom.prabi.fr/prodom/current/html/home.php)
ProDom è un insieme completo delle famiglie dei domini delle proteine generate
automaticamente dalle basi di dati di sequenza di TrEMBL e di SWISS-PROT.
RZPD (http://www.rzpd.de)
È un centro tedesco di risorse per lo studio e la ricerca del genoma.
GENE3D (http://cathwww.biochem.ucl.ac.uk:8080/Gene3D)
Permette di studiare la struttura e le funzionalità delle famiglie di proteine.