O r i g i n a l · Graves-Basedow existe una expresión aumentada de MIP-1α , MIP-1β , MCP-1 y...
Transcript of O r i g i n a l · Graves-Basedow existe una expresión aumentada de MIP-1α , MIP-1β , MCP-1 y...

184
O r i g i n a lVOL. 20 / NÚM. 4 / OCTUBRE-DICIEMBRE 2001
INMUNOLOGÍA, 2001; PP 184-195
Determinación semicuantitativa de la expresión de ARNm de las isoformas de MIP-1α y MIP-1β
mediante un competidor recombinante y RT-AFLP
P. ADREANI1, I. GÓMEZ
1, R. COLOBRAN1, P. CARO, F. PELUSA, R. PUJOL-BORRELL, M. JUAN
Laboratori d’Immunobiologia per a la Recerca i les Aplicacions Diagnòstiques. Centre de Transfusió iBanc de Teixits. Hospital Universitari Germans Trias i Pujol. Universitat Autónoma de Barcelona.
Badalona
RESUMEND e n t ro de la amplia familia de las citocinas quimiotácti-
cas, MIP-1α y MIP-1β son β-quimiocinas de especial inte-rés por sus efectos sobre linfocitos y monocitos, las princi-pales células de la inmunidad adquirida. En estudios pre v i o shallamos una correlación clara entre los niveles tisulares deM I P - 1α y MIP-1β y el diagnóstico clínico en los diversosc u a d ros de enfermedad autoinmune del tiroides. La exis-tencia de isotipos para estas quimiocinas impone cierta difi-cultad en su análisis y evaluación funcional. En este trabajop resentamos un protocolo que, aprovechando pequeñasd i f e rencias a nivel de secuencia nucleotídica, combina RT-PCR y restricción con MspI para estimar semicuantitativa-mente los niveles de ARNm de los loci codificantes para cadauna de estas dos quimiocinas. El aspecto más relevante deesta técnica es el uso de un fragmento de ADN re c o m b i n a n-te que actúa de estándar interno tanto para MIP-1α c o m opara MIP-1β, a la vez que contiene un lugar de re s t r i c c i ó npara MspI. De esta forma actúa como control no sólo deamplificación (en una RT-PCR competitiva) sino tambiénde eficiencia de la digestión. La validez técnica de la apro x i-mación, se corroboró por la similar cinética de amplifica-ción del estándar interno y de las dos citocinas mediantePCR en tiempo real. Un estudio de la inducción de la expre-sión de MIP-1β (mediante experimentos de tiempo-re s-puesta) en células U-937 demostró diferencias claras entrelos ARNms de los dos loci indicando que este protocolo pue-de contribuir al estudio de la expresión diferencial de pro-ductos génicos similares mediante técnicas disponibles enla mayoría de los laboratorios.
PALABRAS CLAVE: MIP-1β/ MIP-1α/ Quimiocinas/ RT-PCR semicuantitativa/ Estándar interno.
ABSTRACTAmong the broad family of chemotactic cytokines, MIP-1α
and MIP-1β , β-chemokines are of special interest, because oftheir central role as chemoattractants for lymphocytes andm a c rophages, the main cells of the adaptive immune re s p o n s e .In previous studies, we have found a relationship between tissuelevels of MIP-1α and MIP-1β and clinical diagnosis in autoim-mune thyroid glands. The measurement of these chemokines isd i fficult because of the existence of diff e rent isotypic forms. Here ,we present a protocol that, taking advantage of small diff e re n-ces in nucleotide sequence, combines RT-PCR and re s t r i c t i o nwith MspI to allow the semiquantitative assessment of mRNAsf rom the two chemokines. The key feature of our approach is thei n t roduction of an internal standard common to MIP-1α a n dM I P - 1β which is co-amplified and contains a MspI re s t r i c t i o nsite. This allows the easy control of both the amplification (com-petitive RT-PCR) and the efficiency of the restriction. Real timePCR experiments demonstrated that the kinetics of the amplifi-cations of the internal standard of both chemokines were simi-l a r, thus validating the approach. A time course of MIP-1β i n d u c-tion in U-937 cells showed substantial diff e rences among themRNAs derived from the two loci, an indication that this tech-nique may contribute to study the diff e rential expression of genep roducts of similar sequence using techniques available at mostl a b o r a t o r i e s .
KEY WORDS: Chemokines/ MIP-1 . MIP-1 / Chemokines/Semiquan-titative RT-PCR/ Internal standard.
S E M I Q U A N T I TATIVE ASSESSMENT OF MIP-1α AND MIP-1β mRNA ISOFORM EXPRESSION BY USING A RECOM-BINANT COMPETITOR FRAGMENT AND RT- A F L P.
1Estos tres autores han contribuido de manera equivalente a la obtención de los resultados presentados, por lo que deberían ser considerados
primeros autores independientemente de la posición que ocupan.

INMUNOLOGÍA P. ADREANI ET AL.
INTRODUCCIÓN
Uno de los elementos definitorios de los pro-cesos inflamatorios implicados en la mayorp a rte de las patologías en las que interv i e-ne el sistema inmunitario, es el grado ycomposición del infiltrado leucocitario. La
migración selectiva de los leucocitos, controla tantolos procesos de re c i rculación fisiológica continua delas células del sistema inmunitario, como la llegadamasiva localizada a las zonas lesionadas (inflama-ción). En ambos procesos, re c i rculación e inflama-ción, intervienen principalmente mecanismos deadhesión intercelular y del leucocito con la matrizextracelular (1). En todos estos procesos adhesivosse utilizan como mediadores moleculares las qui-miocinas, citocinas de pequeño peso molecular quepor su afinidad a la matriz extracelular forman gra-dientes de concentración alrededor de las célulasp roductoras. Sus similitudes estructurales y funcio-nales han llevado a la agrupación de todas estaspequeñas proteínas bajo el término estructural des u p e rfamilia de las quimiocinas.
E n t re las características más relevantes de lasquimiocinas, podríamos decir que son pro t e í n a sde pequeño tamaño (60-70 aminoácidos, apro x i-madamente) y que se encuentran altamente re l a-cionadas en estructura (presentan una homologíade al menos un 25%); así son fundamentales sus 2p r i m e ros residuos cisteínicos del extremo N-ter-minal (2) puesto que son los aminoácidos que per-miten subclasificar esta superfamilia en 4 gru p o sprincipales: a) las CXC o α-Quimiocinas que pre-sentan un aminoácido separando estas 2 primerasCisteinas; b) las CC o β- quimiocinas que no pre-sentan aminoácidos adicionales entre ambas cis-teinas [dentro de este grupo, ha sido identificadala llamada 6Ckine/SLC, la cual presenta un patróninusual de 6 cisteinas en lugar de 4 (3)]; c) las XCquimiocinas o γ-quimiocinas, que pierden el pri-mer y tercer residuo de cisteína (4,5) (sólo se hadescrito la linfotactina en este grupo); y d) lasC X3C quimiocinas caracterizadas por sus 3 ami-noácidos intercalados entre las 2 cisteínas “clasifi-cadoras” (6,7) con la fractalcina como re p re s e n-tante único.
En cuanto a sus propiedades biológicas, las qui-miocinas se distinguen por su capacidad de re g u-lar la movilidad celular por medio de la unión are c e p t o res de membrana que presentan una ciert aespecificidad en el reconocimiento y en su distri-bución celular. Su papel en la adhesión es único,ya que modulan de manera muy precisa las fun-ciones adhesivas de las células diana, pro m o v i e n-do la polarización celular (8), la activación fun-cional de ciertas moléculas de adhesión (9) eincluso la inducción de diversos genes efectore sfundamentales en los mecanismos de adhesión(10,11). No hay que olvidar que las quimiocinasd e s a rrollan también efectos más amplios como la
co-estimulación para la proliferación y difere n-ciación celular o la definición de patrones de sín-tesis de otras citocinas (1,12).
Las quimiocinas actúan, de forma específica yselectiva sobre las células dianas, a través de susre c e p t o res de membrana quienes inducen señalesi n t r a c e l u l a res mediante su acoplamiento a pro t e í-nas G (13,14). Hasta la fecha se han identificadouna veintena de estos re c e p t o res (15-16), aunque,de acuerdo con evidencias funcionales, contamoscon la existencia de otros re c e p t o res no definidosmolecularmente aún (17).
Estudios recientes de nuestro grupo (7), Ashhaby cols. in preparation), indican el posible papel delas β-quimiocinas en los fenómenos autoinmunes,cosa que coincide con el hecho de que en esta sub-familia se encuentran las quimiocinas con un mayorefecto localizador de monocitos y linfocitos (18).Mediante la aplicación y optimización de la meto-dología MOPAC (Mixed Oligonucleotides PrimedAmplification of cDNA) (19), hemos podido demos-trar que en glándulas tiroideas de enfermos deGraves-Basedow existe una expresión aumentadade MIP-1α, MIP-1β, MCP-1 y RANTES, mientrasque no se detecta MCP-3. La especial sobre e x p re-sión de MIP-1α y MIP-1β en las muestras estudia-das re a f i rman datos de otros autores que indican laexistencia de factores de regulación conjunta de lae x p resión de ambas quimiocinas y en concreto enlas enfermedades autoinmunes del tiro i d e s .
N u e s t ro grupo se ha interesado por diversos ele-mentos de la estructura génica del MIP-1β p u e s t oque el genoma humano presenta la secuencia no-alé-lica de un segundo locus. La existencia de dos locipara MIP-1β ya había sido postulada por trabajosp revios (20, 21) donde se les dió el nombre de locus744,1 (al que llamaremos en este artículo locus Acon la idea de simplificar la terminología) y locus744,2 (locus B). Aquellos datos basados en estudiosgenómicos llevaron a sus autores a sugerir que estesegundo locus podía ser un pseudogén (20), aunquelos trabajos de nuestro grupo (7) indican que el locusB puede ser codificante. De hecho, esta duplicidadde loci codificantes para MIP-1β ha sido constadatambién para MIP-1α ( S C YA3 y SCYA3L) en la queel “segundo locus” (SCYA3L, al que también deno-m i n a remos a partir de ahora locus B) codifica parauna segunda forma de MIP-1α ( M I P - 1αP o LD78β)(22). En este trabajo, se expone la apro x i m a c i ó nmetodológica que nos permite valorar la expre s i ó nde MIP-1α y MIP-1β (tanto del locus A como dellocus B). Esta técnica se ha puesto a punto comomedio para comparar la regulación de la expre s i ó nde los dos loci de ambas quimiocinas, y para deter-minar si contribuyen de forma distinta a los fenóme-nos de localización y de modulación de la re s p u e s t ainmunitaria. Nuestros resultados demuestran la fac-tibilidad de la aproximación, que se fundamenta enel uso de un estándar interno como control en unaRT-PCR competitiva y de la discriminación de cada
185

DETERMINACIÓN SEMICUANTITATIVA DE LA EXPRESIÓN DE ARNm DE LAS ISOFORMAS DE MIP-1α Y MIP-1β VOL. 20 NÚM. 4 / 2001
uno de los loci de interés gracias al uso de una endo-nucleasa de restricción. Esta aproximación metodo-lógica, debe considerarse como una herr a m i e n t asimple pero muy útil para la valoración cuantitativade la expresión de los ARNm de los loci A y B de lasquimiocinas MIP-1α y MIP-1β.
MATERIAL Y MÉTODOS
Cultivo celular
N u e s t ros estudios se han realizado con ARN dela línea monocítica U-937 obtenida de la AT C C(American Tissue Culture Collection). El mediode cultivo utilizado fue RPMI 1640 suplementadocon 10% FCS y 2 mM L-glutamina (los tres pro d u c-tos de Life Technologies, Merelbeke, Bélgica), ade-más de gentamicina 40 mM (Sigma, St. Louis, MO,EE.UU.) y Penicilina 100 UI/ml (Laboratorios Ern ,B a rcelona, España). Todas las incubaciones y cul-tivos se hicieron a 37º C con 5% CO2 / 95% aire.
Los cultivos se re a l i z a ron en frascos de 75 cm2
(Costar, Nalge Nunc International, Dinamarca) ensuspensión. Las estimulaciones celulares conIonomicina (1µg/ml), LPS (0,1µg/ml) y PMA(0,1µg/ml) (Sigma, St. Louis, MO, EE.UU.) se lle-varon a término a tiempos de 0, 3, 6 y 24 h, respec-tivamente. Debido a la inestabilidad intrínseca delos extremos UA 3’ del ARNm de las quimiocinas,a g regamos a los cultivos 10 mg/ml de ciclohexi-mida (Sigma, St. Louis, MO, EE.UU.) 3 h antes dela recolección para aumentar la estabilidad.
Extracción de ARN
El método empleado fue el descrito pre v i a m e n t epor Chomczynsky (23) adaptado ligeramente enn u e s t ro laboratorio. Brevemente, el pellet seco pro-veniente de 5.10
6células cultivadas (y estimuladas),
g u a rdado a –70º C hasta el momento de su utiliza-ción, se homogeneiza agregando 500 µl de soluciónde desnaturalización (tiocianato de guanidina 4M;citrato de sodio 25 mM; 2-β m e rcaptoetanol 0,1M;N - l a u ro i l s a rcosina 0,5%; antifoam 0,5%) 50 µl ace-tato de sodio 2M pH 4 y 500 µl de fenol saturadocon agua, mezclando con vórtex después de la adi-ción de cada uno de los reactivos. Se incuba en hie-lo durante 10 min y se agregan 150 µl de una mez-cla de cloro f o rmo-isoamilalcohol que también seincuba en hielo por 10 min más. Se centrifuga 20min a 10.000 rpm (4º C), para separar la fase acuo-sa de la orgánica, precipitándola seguidamente con1 vol de isopropanol (incubación de 1 h a -70º C) ycentrifugando 30 min a 14.000 rpm (4º C). Tras des-c a rtar el sobrenadante, el pellet se resuspende en300 µl de solución de desnaturalización y 150 µl defenol saturado en agua. Se mezcla con un vórt e x ,a g regando seguidamente 150 ml de una mezcla de
c l o ro f o rmo-isoamilalcohol. Tras incubar 10 min a4ºC, se centrifuga a 10.000 rpm durante 20 min(4ºC), transfiriendo el sobrenadante a otro tubodonde se re p recipita con 1 vol de acetato potásico y2 vols de etanol absoluto (post-centrifugación a14.000 rpm durante 30 min a 4ºC). El pellet obte-nido se lava 3 veces con etanol al 70%, se seca bieny se resuspende en 20 µl de agua de DEPC (H2O destilada tratada 20 h con dietilpiro c a r b o n a t o ,[Genaxis Botechnology, Saint-Cloud Cedex,Francia] y posteriormente autoclavada).
La concentración de ARN se determina pore s p e c t rofotometría a una longitud de onda de 260nm, evaluando también su pureza (relación entrelas lecturas a 260 y 280 nm; este valor debe encon-trarse entre 1,8 y 2,0 para que podamos consideraracceptable la extracción). La integridad del ARN sevalora por comparación interna (y con una mues-tra de ARN de E. Coli) por medio de un gel de aga-rosa teñido con bro m u ro de etidio. Se considera queel ARN está íntegro, cuando las bandas corre s p o n-dientes a los ribosómicos presentan apro x i m a d a-mente una intensidad 2/1 en el ratio 28S/18S.
Tratamiento con DNAsa-I de las preparacionesde ARN
Para eliminar los residuos de ADN genómico, quep u d i e ron haber sido extraídos junto con el ARN, lasmuestras son tratadas de la siguiente manera: 2µl detampón (40mM Tris-HCl pH 7,5; 6mM MgCl2), 2µlDNAasaI (Amersham Pharmacia Biotech.B a rcelona. España), Xml de muestra de ARN y 16-Xµl de H2O destilada. La mezcla se incuba a 37º Cdurante 30 min. Luego se cuantifica nuevamente elARN y se re c o m p rueba su integridad. Finalmente,el ARN se precipita con acetato potásico 3M, glucó-geno 20 mg/ml y 1 volumen de isopropanol, laván-dolo con etanol al 75%. El pellet obtenido se diluyeen agua de DEPC y se conserva a -20ºC.
Retrotranscripción
Se desnaturaliza previamente el ARN (1µg dilui-do en agua hasta 11,8 ml) incubándolo a 68ºCdurante 5 min. Inmediatamente después se colocaa 4º C durante 5 min. La reacción de re t ro t r a n s c r i p-ción se realiza según las condiciones indicadas porel proveedor de la Superscript II (Life Te c h n o l o g i e s ,M e relbeke, Bélgica): 4µl de Tampón de primeracadena (5X), 1µl de dNTPS (2mM), 2µl DTT(0,1M), 2µl Oligo-dT24 (500µM), los 11,8µl delARN desnaturalizado, 0,5µl RNAsin (40U/µl), y0,5µl Superscript-II (200U/µl). La reacción se incu-ba 60 min a 42º C. Para inactivar el proceso las mues-tras se calientan hasta 99º C durante 5 min, re e n-friándolas a 4º C (5 min más). Los 20 µl de re a c c i ó nse diluyen 4 veces en EE, usando 1µl de este ADNc
186

INMUNOLOGÍA P. ADREANI ET AL.
diluido para cada 10 µl de reacción de la PCR. To d o slos reactivos utilizados fueron suministrados porP romega (Madison, WI, USA) a excepción de laSuperscript-II y su tampón.
RT-PCR. Condiciones generales
Las diversas RT-PCR se han desarrollado bajo lassiguientes condiciones generales de amplificación:1ml de tampón de PCR (10X, con MgCl2 15 mM);1µl de mix dNTPs (2mM); 0,5 µl de cebador a n t i s e n-s e (10mµM); 0,5µl cebador antisense (10µM); 2µlde ADNc; 3µl de H2O. Las muestras se someten a loscambios de temperatura controlados por el term o-ciclador (MJ. Research, modelo Mini Cycler), ini-ciando el proceso con un inicio en caliente (H o tS t a rt) a 80º C, tras lo cual se agregan 500 mU de laADN-Polimerasa DynaZyme II (Finnzymes OY-Finlandia) diluidas en 2µl de agua. Posteriorm e n t ese aplica el siguiente programa de ciclación: 30 s a95º C, 30 s a 59º C y 30 s a 72º C, que se repite por35 ciclos con una extensión final de 5 min a 72º C.Las secuencias de los cebadores utilizados para laamplificación de MIP-1α son: Cebador α-S e n s e ⇒5’ TTCCGTCACCTGCTCAGAAT 3’; Cebador α-A n t i s e n s e ⇒ 5’ GAAGAGGTA G C T G T G G A G G -TCAC 3’. Para la amplificación de MIP-1β s o n :Cebador β-S e n s e ⇒ 5’ CGCAACTTTGTG-GTA-G AT TA C TAT 3’; Cebador β-A n t i s e n s e ⇒ 5’ AAA-TA AT G G A A AT G A C A C C TA ATA 3’, todos ellos vali-dadados en trabajos anteriores de nuestrol a b o r a t o r i o .
Normalización del ADNc
Para valorar el estado de la muestra y norm a l i-zar los resultados a un parámetro común, todas lasmuestras fueron amplificadas con cebadores espe-cíficos (Oligo Etc, Wilsonville, OR, USA) para elgen de la enzima de expresión constitutivaGAPDH (glutaraldehido-fosfato-deshidro g e n a s a )diseñados en nuestro laboratorio (24): CebadorSense: 5’ CTTCTTTTGCGTCGCCAG 3’; CebadorA n t i s e n s e: 5’ TCTTCTTTTGCGTCGGCCAG 3’.Para esta amplificación también se realiza un H o tS t a rt a 80º C, seguida por 25 ciclos del siguientep rograma: 30 s a 94º C, 30 s a 63º C y 30 s a 72º C,con un extensión final de 5 min a 72º C. La ampli-ficación se evalua con 5µl del amplímero en un gelde agarosa al 2% agarosa (Estándar media EEO,Ecogen, Barcelona, España).
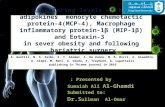
Diseño del Estándar Interno
Para generar el Estándar Interno (EI) se utilizóuna pequeña región del gen de la insulina (zona 3’no codificante), ya que presenta un tamaño (339 pb)
p a recido al de los amplímeros en estudio [MIP-1a(404pb) y MIP-1β (316pb), además de presentar ensu secuencia una diana de restricción para la endo-nucleasa MspI (CCGG). A partir de los cebadore s(ILJ3 e ILJ2) utilizados para estudiar el citado frag-mento del gen de la insulina, se diseñaron dos ceba-d o res híbridos (IEI1 e IEI2) que manteniendo en 3’las secuencias de ILJ3 e ILJ2, se les yuxtapuso con-secutivamente en sus extremos 5’ las secuencias delos cebadores para MIP-1αy para MIP-1β. Para faci-litar su clonación y manipulación, se introdujo tam-bién en estos cebadores híbridos las secuencias dia-na de las enzimas de restricción BamHI y NsiI. (Ve resquema de la Fig. 1), más 2-3 bases en el extre m o5’ para que asegurasen una buena eficiencia en ladigestión. Así, IEI1 es el oligonucleótido re s u l t a n t ede combinar las secuencias de BamHI + β-S e n s e + α-S e n s e + ILJ3, mientras que IEI2 presenta las secuen-cias NsiI + β-A n t i s e n s e + α-A n t i s e n s e + ILJ2 (segúnd i rección 5’-3’ convencional). En la base del esque-ma que se presenta en la figura 1 se detallan lassecuencias concretas de IEI1 e IEI2.
Utilizando los cebadores IE1 e IE2 y las condicio-nes optimizadas para la amplificación del gen de lainsulina con ILJ3 e ILJ2 (35 ciclos del siguiente pro-grama: 20 s a 95º C, 30 s a 65º C y 30 s a 72º C, conun extensión final de 7 min a 72º C) y partiendo de100 ng de ADN genómico humano, se obtuvo una m p l í m e ro de 448 pb. Dicho fragmento se purificópor electroelución en cámara de Biotrap (Schleicher& Schuell; Dassel, Germany) tras electro f o resis ena g a rosa y se clonó por ligación en el vector pZErO-2 (Invitrogen; Paisley, Scotland). Su fácil cre c i m i e n-to en bacterias permite la obtención de un pre p a r a-do homogéneo que una vez cuantificado posibilitasu utilización como estándar interno en una re a c-ción de RT-AFLP (Amplification fragment length poly-m o r p h i s m) competitiva.
PCR Semicuantitativa
Debido a la elevada homología existente entrelos locus A y B de las quimiocinas MIP-1α y MIP-1β a nivel de la secuencia nucleotídica, optamospor estudiar los niveles de expresión de cada unode los loci a través de una RT-PCR competitivasemicuantitativa, a la que asociamos la capacidaddiscriminativa entre loci de los patrones de re s-tricción en base a la endonucleasa MspI. MspI esuna enzima de restricción que reconoce lasecuencia CCGG que en el caso de MIP-1α s eencuentra 3 veces en el producto amplificado dellocus A, pero sólo 1 vez en el locus B; mientrasque en el caso de MIP-1β sólo se encuentra unaúnica vez presente en la secuencia amplificadadel locus A (ninguna en el locus B). Así, de mane-ra secuencial utilizamos la siguiente metodolo-gía: Partiendo de 2 µl de ADNc, se añaden a lareacción 2 µl del Estándar Interno (EI) re c o m b i-
187

DETERMINACIÓN SEMICUANTITATIVA DE LA EXPRESIÓN DE ARNm DE LAS ISOFORMAS DE MIP-1α Y MIP-1β VOL. 20 NÚM. 4 / 2001
nante a concentraciones conocidas (en el mismotubo). Las amplificaciones de MIP-1α y MIP-1β,se realizan sobre muestras separadas utilizandolos oligonucleótidos correspondientes. El pro-ducto así obtenido es sometido a digestión (37ºC durante 1h) en tampón (Y
+/ Tango) con 10 U de
la enzima MspI (Fermentas; Vilnius, Lithuania).P o s t e r i o rmente la definición del patrón de re s-tricción se realiza en gel de agarosa al 2 %. La den-sitometría de las distintas bandas electro f o r é t i-cas permite la cuantificación de las muestrastanto a nivel relativo (pro p o rción de locus A re s-pecto a locus B) como absoluto (en relación conel EI precuantificado). Cabe reseñar aquí, que elEI no sólo permite controlar la amplificación delADNc sino que también a través de su nivel dedigestión (porcentaje de digestión de EI sobre elno digerido) se obtiene otro parámetro básico enesta valoración cuantitativa: el control de ladigestión. Este aspecto es fundamental para laevaluación de MIP-1β, puesto que el locus B, alno presentar diana de restricción para MspI, pue-de confundirse con la parte no digerida del locusA (las digestiones parciales son habituales) y portanto conocer el grado de digestión del EI perm i-te ponderar las cuantificaciones reales de las qui-miocinas de interés.
Real Time PCR
Para comprobar que las cantidades de amplíme-ro producido por la PCR del EI era equiparables conlas que se producen en la amplificación de las qui-
miocinas a estudio, decidimos emplear una apro x i-mación basada en la metodología de PCR a tiemporeal (Real Time PCR o rt-PCR). La monitorizaciónde la rt-PCR se realizó con el fluoro c romo interc a-lante SYBRGreen (Roche; Mannheim, Germ a n y )con el equipo LightCycler® (Roche; Mannheim,G e rmany). Para ello, utilizamos diluciones seriadastanto de amplímeros precuantificados de los loci enestudio, como del EI (realizando el estudio tres vecescon muestras por duplicado). Los ensayos se desa-rro l l a ron bajo los siguientes parámetros: 1µl de lamezcla de reactivos de rt-PCR 10X (Taq ADN poly-merase, reaction buffer dNTP mix [con dUTP enlugar de dTTP], SYBR Green I dye y 10 mM MgCl2) ;1,2 µl de MgCl2 25mM (concentración final 4 mM);0,5 ml de cebador s e n s e (10 µM); 0,5µl cebadora n t i s e n s e (10 µM); 3 µl de molde-muestra; 4,8 µl deH2O. El estudio realizado tanto con MIP-1α c o m ocon MIP-1β, se presenta aquí sólo para MIP-1β.
Las amplificaciones se re a l i z a ron en 35 ciclosdel siguiente programa: 30 s a 95º C, 30 s a 59º C,30 s a 72º C con una extensión final de 5 min a 72º C. Las lecturas de fluorescencia se re a l i z a ro na 72º C en cada ciclo y la evaluación de la tempera-tura de fusión de los productos amplificadosdurante el período final de desnaturalización entre45º C y 95º C.
RESULTADOS
1. Obtención de un estándar interno co-amplifi-cable con el ADNc nativo de MIP-1α y MIP-1β c o m ocontrol de RT-PCR.
188
Gen Insulina 339 pb
MspI
lEl1
BamHI
lEl277 bNsiI
β-asα-as
9 b25 b
23 b20 bILJ2
α-sβ-s
ILJ3 19 b20 b
24 b
71 b8 b
lEl1: CGGGATCC CGCAACTTTGTGGTAGATTACTAT TTCCGTCACCTGCTCAGAAT TGGAGATGGGTGGGAGTGT
lEl2: CCAATGCAT AAATAATGGAAATGACACCTAATAC GAAGAGGTAGCTGTGGAGGTCAC AGGGCTTTATTCCAT
BamHI β-sense α-sense ILJ3
NsiI β-antisense α-antisense ILJ2
Figura 1. Esquema del estándar interno(EI) utilizado en nuestra aproximación de RT-PCR. En línea gruesa, se pre s e n-ta el amplímero del gen de la insulina con el punto de corte para MspI. En trazo más fino asociado al amplímero del gende la insulina se re p resentan los cebadores híbridos IEI1 e IEI2, cuya secuencia concreta se precisa en la base del esque-ma señalando los nucleótidos correspondientes a las secuencias de los cebadores de la insulina (ILJ3 y ILJ2), MIP-1α (α-sense y α-antisense) y MIP-1β(β-sense y β-antisense) además de las dianas de restricción (BamH1 y NsiI). Las abre v i a-turas b y pb se re f i e ren las longitudes de las diversas regiones expresadas en bases y pares de bases re s p e c t i v a m e n t e .

INMUNOLOGÍA P. ADREANI ET AL.
La necesidad de controlar el proceso de ampli-ficación de los loci de MIP-1α y MIP-1β, nos lle-vó a diseñar unos cebadores híbridos que tras laamplificación de un gen no-relacionado nos rin-d i e ron un producto de PCR de distinto pesomolecular al de los amplímeros en estudio, peroque contenía en sus extremos las secuencias delos cebadores que se pretenden utilizar paraamplificar el ADNc tanto de MIP-α como de MIP-1β (ver Fig. 1). En el caso de MIP-1α (Fig. 2A),los productos amplificados en esta RT-PCR com-petitiva con los cebadores α-sense y α- a n t i s e n s ese distinguen por su peso molecular, ya que mien-tras el amplímero del gen rinde un producto de404-407 pb (404 pb según la secuencia de locusA y 407 pb para el locus B), el producto del ampli-ficado con el EI es de 382 pb (menor peso mole-cular que el gen de interés). En cambio para MIP-1β (Fig. 2B), la RT-PCR competitiva con losc e b a d o res β-sense y β-antisense produce sobre elEI un amplímero de mayor peso molecular (431pb) que el del MIP-1β (316 pb tanto para locus Acomo para locus B). Cabe destacar que en amboscasos es demostrable durante la reacción deamplificación la competición entre el ADNc deinterés y el ADN del EI.
2. La PCR competitiva permite la cuantificaciónen número de copias del gen de interés.
P a rtiendo de ADNcs, normalizados para GAPDH,la PCR competitiva permite deducir el número decopias iniciales (de MIP-1α o MIP-1β) en el ADNcp roblema, a partir de un número de copias conoci-das en el EI coamplificado. La figura 3 muestra unejemplo con una de estas cuantificaciones para MIP-1β, en una muestra proviniente de células U-937estimuladas con LPS y tratadas con cicloheximida.De hecho la metodología atribuye el número decopias iniciales que la PCR amplifica por compara-ción con el número de copias iniciales del EI. Así, ensentido estricto, sólo si la cantidad del amplímeroobtenido de MIP-1β (que incluye las amplificacio-nes de locus A y de locus B) tuviera su concentra-ción molar igual a la del EI, podríamos deducir que
189
A. Co-amplificación de E.I.( ) y MIP-1α (→)
B. Co-amplificación de E.I.( ) y MIP-1β ( )
–
cDNA
E.I. 103
cDNA
–
–
Phix174RF/Rsal
E.I. 103
cDNA
E.I. 104
cDNA
E.I. 105
cDNA
Phix174RF/Rsal
→
Figura 2. Comparación de las co-amplificaciones deEI con MIP-1α o MIP-1β de U-937 estimuladas. En Ase muestra la co-amplificación del estándar Intern o(EI) y el ADNc de MIP-1α donde la banda superiorc o rresponde al amplímero de MIP-1α (404 pb) y labanda inferior al EI (382 pb). En B se muestra la coam-plificación del EI y el ADNc de MIP-1β, donde la bandasuperior corresponde al EI (431 pb) mientras que labanda inferior corresponde al amplímero del ADNc deM I P - 1β (316 pb).
X
X
104 103105
X E.I
cDNA
30.000
20.000
10.000
40.000
X
Copias
Figura 3. Cuantificación de la reacción competitiva. Enel lado izquierdo de la figura se muestra la electro f o resis engel de agarosa de una coamplificación del Estándar Intern o(EI) a concentraciones decrecientes (10
5, 10
4y 10
3) y del
ADNc de las muestras de MIP-1β de U-937 estimuladas(cantidad fija). En la zona izquierda del lado derecho de lafigura, se muestra la graficación lineal de esta amplifica-ción, para extrapolar y cuantificar la cantidad de MIP-1βtotal de la muestra problema. Para ello se grafican (demanera logarítmica) las copias iniciales del EI respecto alas densidades ópticas de sus amplímeros electro f o r é t i c a-mente resueltos (unidades arbitrarias) y densitométrica-mente evaluados (X de color gris). A partir del valor den-sitométrico del ADNc (X de color negro) de la muestra pro-blema (en su dilución más equivalente a la amplificacióndel EI), se extrapola de la gráfica el número de copias ini-ciales de la muestra.
E.I.
cDNA10
5
+10
4
+
E.I.
cDNA
103
+

DETERMINACIÓN SEMICUANTITATIVA DE LA EXPRESIÓN DE ARNm DE LAS ISOFORMAS DE MIP-1α Y MIP-1β VOL. 20 NÚM. 4 / 2001
la concentración inicial también era idéntica.Cuando los valores no son idénticos (lo habitual),p rocedemos a graficar la relación que existe entre elEI y la muestra en la amplificación más semejante(se grafican los valores densitométricos de las cita-das bandas electroforéticas o si se introduce marc a-je radioactivo la señal radioactiva de dichos amplí-m e ros) tal y como se muestra en la figura 3,deduciendo a partir de la gráfica el número de copiasen el problema. En cualquier caso, la realización delcálculo de ratios en pro p o rción logarítmica (25) per-mite la cuantificación más exacta de los pro d u c t o sa m p l i f i c a d o s .
3. El estándar interno actúa como control de ladigestión para la evaluación por AFLP posterior dela pro p o rción de los loci A y B de MIP-1α y M I P - 1β.
El protocolo de co-amplificación pro p u e s t op e rmite, a través de la digestión con MspI, dis-tinguir el nivel de expresión de los loci A y B tan-to de MIP-1α como de MIP-1β (Fig. 4). Dado queel EI coamplificado presenta una diana de re s t r i c-ción, este estándar nos permite monitorizar estadigestión al liberar dos fragmentos de re s t r i c c i ó nde distinto peso molecular (Tabla I). Así tras laamplificación del ADNc y el EI con los cebado-res de MIP-1α tal y como se ha comentado, seobtienen dos amplímeros de 404-407 pb por par-te de MIP-1α (404 pb según la secuencia de locusA y 407 pb para el locus B) y de 382 pb por part edel EI. La digestión con MspI digiere los 382 pbdel EI en dos fragmentos de 265 y 117 pb que sonp e rfectamente distinguibles de la longitud de losfragmentos de restricción procedentes de MIP-1α: 176, 138, 62 y 28 pb por parte del Locus A (4bandas) y 231 y 176 pb por parte del locus B (2bandas). Para la amplificación del ADNc y el EIcon los cebadores de MIP-1α, se obtienen una m p l í m e ro de 431 pb por parte del EI y otro de316 pb por parte de MIP-1b. En este caso (Fig. 4),la digestión con MspI produce fragmentos de 290y 141 pb provenientes del EI y de 316 (locus B),190 y 126 pb (correspondientes al locus A) porlo que se re f i e re a MIP-1β. De hecho es en el estu-dio de MIP-1β donde se demuestra la mayor uti-lidad del EI como control de la digestión conMspI, puesto que debido a que la valoración dellocus B es sólo posible por la ausencia de la dia-na de MspI en el locus B, es imprescindible unbuen control que permita valorar el nivel dedigestión parcial de la misma. Este nivel de diges-tión, calculado como porcentaje de la densidadrestante de la banda de 431 pb respecto a la de290 pb ponderada por 1,48 (la relación entre las431 bases y las 290 de ambas bandas), nos per-mite re e v a l u a r, con un mayor nivel de pre c i s i ó n ,la cuantificación densitométrica del locus B, evi-tando que se sobre v a l o re su expresión por la pro-p o rción de locus A no digerido y a su vez la dellocus A que si no, se vería subvalorado.
190
Estándar Interno(431 pb)
cDNALocus A + Locus B(316 pb)
Digestión parcial E.I.
Locus B (315 pb)
Fragmento 1 Locus A(190 pb)
Fragmento 2 EstándarInterno (141 pb)
Fragmento 2 Locus A(126 pb)
NoDigestivo
Digeridocon Mspl
Fragmento 1 EstándarInterno (290 pb)
Figura 4. Discriminación de los isotipos de MIP-1β.La figura muestra la resolución electroforética de ladigestión (locus A del MIP-1β y el Estándar Intern o(EI)) con la enzima de restricción MspI en relación conla misma muestra sin digerir. El carril izquierdo mues-tra el ADNc de MIP-1β y del EI sin digerir, definiéndo-se 2 bandas: una de 431 pb (correspondiente al EI) y laotra de 316 pb (que corresponde a los ADNc del locus Ay del locus B). El carril derecho muestra la digestión dela misma muestra por la enzima MspI, la cual origina5 bandas: 2 que corresponden al EI (290 pb y 141 pb),2 al ADNc del locus A (190 pb y 126 pb) y una al ADNcdel locus B (315 pb) que no es digerible por la enzima.Además se señala con una flecha superior los restos deEI que permanecen por falta de digestión (digestiónp a rc i a l ) .
MIP-1α MIP-1βDigestión Mspl - + - +
Moldes para PCR
265 290
EI 382 431
117 141
407 (B) 231 (B)
176 (A y B) 316 (B) 316 (B)
cDNA 138 (A) 316 (A) 190 (A)
404 (A) 62 (A) 126 (A)
28 (A)
En la tabla se muestran los pesos moleculares (en pares de bases) de los pro d u c-tos de amplificación con los cebadores específicos para MIP-1α y MIP-1β y de lossubsiguientes fragmentos de restricción obtenidos tras la digestión con Mspl. Enla tabla se separan los productos del EI y de los genes de interés (el molde usadoson los cDNA obtenidos tras re t rotranscripción de los RNA´s extraidos de losmuestras a estudio) aunque en la metodología propuesta los fragmentos de unoy otro se entremezclan en la electro f o resis convencional sobre gel de agaro s a .E n t re paréntesis se expresan los loci génicos de los que provienen los amplíme-ros y fragmentos discriminados. Las flechas indican la procedencia de los frag-m e n t o s .
Tabla ILongitud (en pb) de los productos de amplifi-cación y de los fragmentos de restricción tras
RT-AFLP para MIP-1α y MIP-1β

INMUNOLOGÍA P. ADREANI ET AL.
4. Uso de la 1a RT-AFLP para la evaluación de lainducción génica de MIP-1α y MIP-1β.
A partir de la cuantificación densitométrica de losfragmentos de restricción con MspI, se determ i n asemicuantitativamente el número de copias trascri-tas por el locus A o el locus B de ambas β- q u i m i o c i-nas, obteniendo a su vez una información exacta dela pro p o rción en que ambos loci contribuyen a la sín-tesis de cada quimiocina. En la figura 5 se muestracomo esta estrategia aplicada al estudio de MIP-1βp e rmite discriminar la inducción diferenciable entreel locus A y el locus B frente a diversos estímulos ytipos celulares. La figura 5A ejemplifica en uno delos geles para el estudio de la expresión de MIP-1βen la línea mielomonocítica U-937 frente a la esti-mulación con Ionomicina, LPS y PMA. En la figura5B, se grafican los resultados densitométricos de esteexperimento una vez analizados y ponderados, dis-
tinguiendo la contribución a la inducción de lae x p resión de los loci de MIP-1β. De hecho, tanto estafigura de ejemplo como otros datos propios (en pre-paración para su publicación) realizados con estaestrategia nos han permitido demostrar que los estí-mulos inductores actúan a niveles y con cinéticasmuy distintos para locus A y locus B de MIP-1β l ocual implica diferencias en sus pro m o t o res, que has-ta ahora no han sido estudiados de forma compara-tiva. De manera equivalente, la metodología es apli-cable al estudio de los locus A y B de MIP-1α.
5. Comparacación de las cinéticas de la reacción deamplificación del EI con la de los ADNcs de MIP-1β.Evaluación conjunta de la cinética de co-amplificación.
Dado que la metodología de la co-amplificacióncompetitiva respecto a un estándar pre c u a n t i f i c a d op resupone que el comportamiento del estándar esequivalente al del gen que se estudia, planteamos
191
12000
8000
4000
00h 3h 6h 24h
1. Cuantificación de la expresiónde MIP-1β Locus A
2. Cuantificación de la expresiónde MIP-1β Locus B
0h 3h 6h 24h
900
600
300
0
PMA LPS Ionom. No estim.
Figura 5. Análisis semicuantitativo de MIP-1β tras la estimulación de células U-937. En A se muestra la resolución elec-t roforética de la cinética (situación basal, 3h, 6h y 24h post-estímulo) de inducción de la expresión tanto del locus A como dellocus B del MIP-1β tras la estimulación de la línea celular U-937 con PMA, LPS e Ionomicina. También se muestra la diges-tión (+) y la no digestión (-) con la enzima MspI en cada uno de los casos. Las muestras fueron normalizadas previamente conGAPDH (imagen inferior de la figura). En B se re p resentan las cuantificaciones normalizadas de la expresión del MIP-1β t a n t odel locus A (lado izquierdo) como del locus B (lado derecho) tras las citadas estimulaciones con PMA, LPS e Ionomicina.
+ - + - + - + -+ - + - + - - + - +- + - +
PMA
Dig.Mspl
Noestim.3h 6h 24h 3h 6h 24h
GAPDH
MIP-1βLPS lonomicina
3h 24h6h
B
A

DETERMINACIÓN SEMICUANTITATIVA DE LA EXPRESIÓN DE ARNm DE LAS ISOFORMAS DE MIP-1α Y MIP-1β VOL. 20 NÚM. 4 / 2001
analizar la veracidad de esta afirmación medianteel uso de la PCR en tiempo real (rt-PCR). Para ello,comparamos las cinéticas de amplificación de losADNcs de los genes a estudio respecto al EI, median-te la monitorización continuada que ofrece la rt -PCR. Concretamente, la figura 6 muestra las curv a sde amplificación para los cebadores de MIP-1β a p l i-cados a amplímeros de MIP-1β ( p recuantificados ydiluidos hasta 10
7y 10
6copias iniciales) en re l a c i ó n
a las curvas del EI que hemos desarrollado (pre-cuantificados y diluidos a 3x10
7, 10
6y 10
5c o p i a s
iniciales). Las curvas de amplificación del EI, si bienno son idénticas a las propias del MIP-1β, sí que songlobalmente equiparables, en especial las pendien-tes de las mismas que definen la eficiencia de ampli-ficación y por tanto la cantidad de copias del moldede partida. De hecho, el estudio demostró que estasimilitud de comportamiento se produce especial-mente en el rango de 10
7- 1 0
5copias iniciales. Por
debajo y por encima de ello existe una mayor diver-gencia entre el EI y el producto normal (datos nomostrados). En cualquier caso, estos resultados nosdemuestran que el EI puede considerarse una apro-ximación absolutamente aceptable en la evaluaciónsemicuantitativa de la expresión de los transcritosde los genes de MIP-1β.
DISCUSIÓN
Los resultados presentados muestran que eld e s a rrollo y uso del fragmento competidorrecombinante (EI) permite la valoración semi-
cuantitativa por RT-PCR del ARNm de las qui-miocinas MIP-1α y MIP-1β, de manera sencilla yfiable. Conceptualmente, el interés real de estaa p roximación radica en el estudio de las funcio-nes de las quimiocinas, al quedar bien estableci-do que su expresión define la localización de lascélulas inmunitarias y, por tanto, el funciona-miento del sistema inmunitario (26) y de losó rganos linfoides (27-28), en situaciones fisioló-gicas y patológicas (29-30). Su valoración entodas estas condiciones, a menudo re q u i e re de laevaluación de los ácidos nucleicos codificantes.La cuantificación de los transcritos es necesariaen las quimiocinas inducibles, como MIP-1α yM I P - 1β, en las que se conoce que los cambiosrelativos se relacionan con comport a m i e n t o sfuncionales distintos (31). El uso de la RT- P C Rsin duda posibilita esta evaluación, aunque ladiversidad de comportamiento de la PCR en lasdiversas circunstancias de los ensayos y su ciné-tica que converge hacia la meseta de máximaamplificación hacen necesario el uso de contro-les del proceso. Existen diversas apro x i m a c i o n e spara controlar el proceso, pero hasta la re c i e n t ei rrupción de la PCR en tiempo real, el uso de con-t roles internos (en cada tubo de amplificación)se ha demostrado como la mejor apro x i m a c i ó nmetodológica.
La existencia de 4 loci genéticos codificantes(locus A y locus B) de MIP-1α y MIP-1β obliga adiseñar estrategias para su análisis por separado sise desea estudiar su contribución individual al con-junto. Dada la gran similitud entre estas isoform a s
192
Cycle Number
35,0
32,5
30,0
27,5
25,0
22,5
20,0
17,5
15,0
12,5
10,0
7,5
5,0
2,5
0,0
-2,55 100 15 20 30 3525 40 45 50 55 60 65
3x107 EI
107 MIP-1β
106 EI
106 MIP-1β
105 MIP-1β
Figura 6. Comparación de cinéticas de amplificación del EI o fragmento competidor respecto al ADNc de MIP-1β. Sep resentan las curvas obtenidas por rt-PCR (PCR a tiempo real) que muestran cinéticas de amplificación tanto del MIP-1βcomo del EI. Las curvas corresponden a las amplificaciones de las diferentes diluciones del EI (3x107, 106, y 105 copias), yde amplímeros pre-cuantificados de MIP-1β ( 1 07, y 105 copias). Merece la pena observar que las cinéticas, si bien no sonidénticas, sí que pueden considerarse equivalentes, en especial respecto a las pendientes de amplificación de las mismas.

INMUNOLOGÍA P. ADREANI ET AL.
(los mensajeros no sólo son prácticamente igualesen cuanto a longitud, sino que incluso las secuen-cias son más de un 99% homólogas), este estudiore q u i e re la combinación de otra metodología quese añade a la simple RT-PCR. En nuestro caso, plan-teamos como estrategia metodológica el uso deMspI que posibilita la discriminación de ambos locipor la longitud de los fragmentos de restricción delos amplímeros (RT-AFLP). Para poder cuantifi-carlos, hay que controlar los dos parámetros quepodrían introducir más variaciones en este doblep roceso: la diversidad de la cinética de amplifica-ción del ADNc y la eficiencia de la digestión conMspI. Con este objetivo, de nuevo el EI será unah e rramienta imprescindible puesto que permite a)c o n t rolar la eficiencia de la digestión con la citadaendonucleasa sobre cada tubo, distinguiendo losfragmentos no digeridos (no presentan esta diana),y b) recuantificar de manera precisa los valores cal-culados por densitometría. De hecho en el casoc o n c reto de MIP-1β, los loci A y B presentan unahomología muy elevada en su secuencia codifican-te (ADNc), aunque difieren en algunas bases. Estad i f e rencia es clave para la identificación de cadalocus al utilizar la enzima MspI que reconoce lasecuencia CCGG, presente sólo en el locus A (ellocus B presenta la secuencia CTGG). Cuando sep roduce el corte, se generan 2 fragmentos en ellocus A; mientras que el locus B queda intacto.Estos fragmentos son perfectamente visualizablesen un gel de agarosa. Merece la pena destacar aquí,que en algunos casos también se puede distinguiruna forma truncada del locus B, que presenta 15 pbmenos (datos no mostrados).
Esta aproximación metodológica se demuestrasimple y útil para el estudio de la expresión de lasquimiocinas MIP-1α y MIP-1β, aunque pre s e n t aalgunas limitaciones que podrían cuestionar suexactitud. Por un lado, tal y como se describe, setrata de una estrategia que más que cuantitativadebe adjetivarse como semi-cuantitativa (y así secataloga a lo largo del trabajo) El prefijo semi- debeaplicarse porque los valores de copias iniciales detranscrito no son absolutos, ya que no se corre l a-cionan exactamente con los ARNm de partida. Elc o n t rol que aporta el EI se aplica a partir del ADNc,por lo que no monitoriza los procesos pre v i o s(específicamente la extracción de ARN desde lascélulas y la posterior re t rotranscripción). Si bienno resulta demasiado atrevido asumir eficienciasequivalentes para estos procesos previos a la ampli-ficación, en sentido estricto sería convenientei n t roducir el control desde la extracción. Para ellobastaría con transcribir el EI in vitro a partir del plás-mido recombinante (aprovechando para ello lasecuencia promotora Sp6 del pZErO-2), cuantifi-car el ARN producido y añadirlo a la concentraciónadecuada en el mismo momento de la extracción.De este modo, este EI en forma de ARNm contro-laría la eficiencia de todo el proceso. Es obvio que
esta estrategia aunque más precisa, resulta técni-camente más compleja y delicada, y si bien puedeser requerida en determinadas aplicaciones (porejemplo evaluación de cargas virales para viru sARN), en general no suele exigirse en el estudio dela expresión de genes eucariotas, asumiendo quela aplicación de un control a partir de la PCR per-mite obtener resultados que “sólo pueden consi-derarse semicuantitativos” (como es nuestro caso).En este sentido, el uso de un EI competidor (inclu-so en forma de ARN) de distinto peso molecularp resupone que éste mantiene la misma eficienciade amplificación que el ADNc de interés. Y de nue-vo esta presunción, si bien en general es aceptable,en sentido estricto es como mínimo imprecisa. Dehecho, nuestros resultados (Fig. 6) demuestranesta imprecisión, puesto que las diferencias desecuencia de los moldes (en este caso debido al dis-tinto peso molecular) influyen en la eficiencia dela amplificación. Por ello, se diseñan EI de pesomolecular muy similar al del producto a estudio,p refiriendo usar en general, estándares internos demayor peso molecular (como MIP-1β) que demenor peso (como MIP-1α). Con ello, se buscas o b reestimar el gen de interés (en general los amplí-m e ros de menor peso molecular se amplificanmejor), evitando la infravaloración que un EImenor que el gen de interés pudiera producir alcompetir con mayor eficiencia.
Todas estas limitaciones conceptualmente seevitan con la aplicación de la RT-PCR a tiempo re a l ,puesto que el seguimiento continuo de la re a c c i ó nque permiten estos equipos hace innecesaria lai n t roducción de un EI de distinto peso en la mues-tra. En la rt-PCR, son los parámetros de la cinéticade amplificación (pendiente, señal de fondo, mese-ta final, etc.) los que permiten asegurar la cuantifi-cación por comparación respecto a muestras pre-viamente cuantificadas que se amplifican enreacciones simultáneas (e incluso pre v i a m e n t ealmacenadas). Es posible que en aras de una máxi-ma precisión fuera aconsejable cuantificar global-mente MIP-1α o MIP-1β por rt-PCR, añadiendo aposteriori el EI amplificado para que controlara elnivel de eficacia de la digestión, perm i t i é n d o n o sasí definir la contribución de cada loci a esta canti-dad global de CC-quimiocinas. En cualquier caso,n u e s t ros resultados con rt-PCR, confirman que laa p roximación más clásica con el EI parece pro p o r-cionar resultados globalmente equiparables a losde la rt-PCR y por lo tanto igualmente aceptables,lo cual sin duda es importante dado que los equi-pos de PCR en tiempo real todavía no están dispo-nibles más que en contados centros. El desarro l l op ropuesto se ha diseñado para evaluar pre f e re n t e-mente la expresión de los loci A y B de MIP-1β, másque de los correspondientes de MIP-1α. De hecholos loci de MIP-1β son mucho más homólogose n t re ellos que las secuencias de las isoformas deM I P - 1α, para las que otras estrategias más simples
193

DETERMINACIÓN SEMICUANTITATIVA DE LA EXPRESIÓN DE ARNm DE LAS ISOFORMAS DE MIP-1α Y MIP-1β VOL. 20 NÚM. 4 / 2001
194
pueden ser tanto o más útiles. En cualquier caso,este artículo no pretende definir las peculiaridadesde regulación de ambos loci de MIP-1β, sino ilus-trar la fiabilidad de la metodología desarro l l a d a .Así pues, en resumen, este trabajo permite afirm a rque a través de nuestra aproximación es posiblecuantificar de una manera fácil, rápida y económi-ca la cantidad de mensajero de MIP-1α y MIP-1β,definiéndose además la participación en esta expre-sión de los transcritos codificados por el locus A ylos codificados por el locus B.
AGRADECIMIENTOS
Este trabajo ha podido ser realizado gracias a lafinanciación del Fondo de InvestigacionesSanitarias (FIS99/1063). Agradecemos especial-mente al Dr. Yaqoub Ashhab y al Dr. OrlandoDomínguez, quienes habiendo realizado trabajosprevios en nuestro laboratorio nos abrieron la líneade investigación que ha condicionado y guiado eld e s a rrollo del trabajo presentado en nuestro art í-culo. También quisiéramos extender nuestro agra-decimiento a Pilar Armengol, por su orientacióndurante el desarrollo del trabajo, así como supaciencia y dedicación.
Bibliografía1. Springer TA. Tr a ffic signals for lymphocyte re c i rc u l a-
tion and leukocyte emigration: The multistep para-digm. Cell 1994; 76: 301-14.
2. Zlotnik A, Yoshie, O. Chemokines: a new classificationsystem and their role in immunity. Immunity 2000; 12:121-127.
3. Hedrick JA, Zlotnik A. Identification and characteriza-tion of a novel beta chemokine containing six conser-ved cysteines. J Immunol. 1997; 159: 1589-93.
4. Kennedy J, Kelner GS, Kleyensteuber S, Schall TJ,Weiss MC, Yssel H et al. Molecular cloning and functio-nal characterization of human lymphotactin. JImmunol 1995; 155: 203-9.
5. Kelner GS, Kennedy J, Bacon KB, Kleyensteuber S,L a rgaespada DA, Jenkins NA, et al. Lymphotactin: acytokine that re p resents a new class of chemokine.Science 1994; 266: 1395-9.
6. Pan Y IC, Zhou H, Dolich S, Deeds J, Gonzalo JA, Vath J etal. Neurotactin, a membrane-anchored chemokine upre-gulated in brain inflamation. Nature 1997; 387: 611-7.
7. Ashhab Y. β-Chemokines in thyroid autoimmunity.Tesis doctoral UAB 1998.
8. del Pozo MA, Sánchez-Mateos P, Sánchez-Madrid F.Cellular polarization induced by chemokines: a mecha-nism for leukocyte re c ruitment? Immunol Today 1996;17: 127-31.
9. C a rr MW, Alon R, Springer TA. The C-C chemokine MCP-1 diff e rentially modulates the avidity of beta 1 and beta 2integrins on t lymphocytes. Immunity 1996; 4: 179-87.
10. Wu L, Fan J, Matsumoto Si, Watanabe T. Induction andregulation of matrix metalloproteinase-12 by cytokinesand CD40 signaling in monocyte/macrophages. BiochemByophys Res Commun 2000; 269: 808-15.
11. Yamamoyo T, Eckes B, Mauch C, Hartmann K, Krieg T.Monocyte chemoattractant protein-1 enhances genee x p ression and synthesis of matrix metallopro t e i n a s e - 1in human fibroblasts by an autocrine IL-1α loop. J Immunol 2000; 164: 6174-9.
12. Lindley JD, Kunkel SL. Nomenclature announcement:the chemokines. Immunol Today 1993; 14: 24.
13. Murphy PM. The molecular biology of leucocytes che-moattractant receptors. Ann. Rev. Immunol 1994; 12:593-633.
14. B e n - B a ruch A, Michiel, DF, Oppenheim J. Signal andreceptors involved in recruitment of inflamatory cells. JBiol Chem 1995; 270: 2411-4.
15. Power CA CD, Meyer A, Alouani S, Proudfoot AE,Clark-Lewis I, Sozzani S, et al. Cloning and characteri-zation of a specific receptor for the novel CC chemoki-ne MIP-3alpha from lung dendritic cells. J Exp Med1997; 186: 825-35.
16. Wells TN, Lusti-Narasimhan M, Chung CW, Cooke R,Power CA, Peitsch MC, et al. The molecular basis ofselectivity between CC and CXC chemokines: the pos-sibility of chemokine antagonists as anti-inflamatoryagents. Ann N Y Acad Sci 1996; 796: 245-56.
17. G reaves DR. Chemokines-the class of ’97. ImmunologyNews 1997; 4: 257-9.
18. Baggiolini M, Moser B. Interleukin-8 and related che-motactic cytokines CXC and CC chemokines. Advancein Immunology 1994; 55: 97-179.
19. Ashhab Y, Domínguez O, Sospedra M, Roura-Mir C,Lucas-Martín A, Pujol-Borrell R. A one-tuve polymerasechain reaction protocol demonstrates CC chemokineo v e re x p ression in Graves’ Disease Glands. J ClinEndocrinol Metab 1999; 84: 2873-82
20. Napolitano M, Modi WS, Cevario SJ, Gnarra JR,Seuanez HN, Leohard WJ. The gene encoding the Act-2cytokine. J. Biol. Chem 1991; 266: 17531-6.
21. I rving S ZP, Balke J, McBride O, Morton C, Burd P,Siebenlist U, et al. Two inflamatory mediador cytokinegenes are closely and variably amplified on chro m o -some 17q. Nucleic Acid Research 1990; 18: 3261-70.
22. Nibbs R, Yang J, Landau N, Mao J, Graham G. LD78β, anon-allelic variant of human MIP-1α ( L D 7 8α), hasenhanced receptor interactions and potent HIVSuppressive Activity. J Biol Chem 1999, 274: 17478-83.
23. Chomczynski P, Sacchi N. Single step method of RNAIsolation by acid guanidinium thioccyanate-phenol-cloro-f o rm extraction. Analytical Biochemestry 1987; 162: 156-9.
24. Sospedra M, Ferre r-Francesch X, Domínguez O, JuanM, Foz-Sala M, Pujol-Borrel R. Transcription of a broadrange of self-antigens in human thymus suggests a ro l efor central mechanisms in tolerance toward peripheralantigens. J Inmunol 1998; 161: 5918-29.
25. Bloch W. A biochemical perspective of the polymerasechain reaction. Biochemistry 1991; 30: 2735-47.
26. Sallusto F, Lenig D, Forster R, Lipp M, Lanzavecchia A.Two subsets of memory T lymphocytes with distincthoming potentials and effector functions. Nature 1999;401: 708-12.
27. Bowman EP, Campbell JJ, Soler D, Dong Z, ManlongatN, Picarella D, et al. Developmental switches in chemo-kine response profiles during B cell diff e rentiation andmaturation. J Exp Med 2000; 191(8): 1303-18.
CORRESPONDENCIA:Manel Juan i OteroUnitat d´ Immunologia, Lirad-CTBTHospital Universitari Germans Trias i PujolCtra. Canyet s/n. P.O. BOX 7208916 Badalona (Barcelona). España

INMUNOLOGÍA P. ADREANI ET AL.
195
28. Sallusto F, Lanzavecchia A.Understanding dendritic celland T-lymphocyte traffic through the analysis of che-mokine receptor expression. Immunol Rev 2000; 177:134-40.
29. Shi K, Hayashida K, Kaneko M, Hashimoto J, Tomita T,Lipsky PE, et al. Lymphoid chemokine B cell-attractingchemokine-1 (CXCL13) is expressed in germinal centerof ectopic lymphoid follicles within the synovium ofchronic arthritis patients. J Immunol 2001; 166: 650-5.
30. A rmengol MP, Juan M, Lucas-Martín A, Fern á n d e z -Figueras MT, Jaraquemada D et al. Ectopic lymphoid folli-cles in thyroid autoimmune glands contain active autore a c-tive germinal centres. Am J Pathol 2001; 159: 861-73.
31. C a m e ron MJ, Arreaza GA, Grattan M, Meagher C,Sharif S, Burdick MD, et al. Diff e rential expression ofCC chemokines and the CCR5 receptor in the pancreasis associated with pro g ression to type I diabetes. JImmunol 2000; 165: 1102-10.