IDENTIFICACION Y LOCALIZACIÓN DE SECUENCIAS DE …
Transcript of IDENTIFICACION Y LOCALIZACIÓN DE SECUENCIAS DE …

12
IDENTIFICACION Y LOCALIZACIÓN DE SECUENCIAS DE
REGULACIÓN DE LA EXPRESIÓN DE LA
FAMILIA DE GENES DE β-GLOBINA
EN DIFERENTES ESPECIES
NATALIA IVVON ACEVEDO LUNA
PONTIFICIA UNIVERSIDAD JAVERIANA
FACULTAD DE CIENCIAS
CARRERA DE BIOLOGIA
Bogota, D.C
Junio, 2007

13
IDENTIFICACION Y LOCALIZACIÓN DE SECUENCIAS DE
REGULACIÓN DE LA EXPRESIÓN DE LA
FAMILIA DE GENES DE β-GLOBINA
EN DIFERENTES ESPECIES
NATALIA IVVON ACEVEDO LUNA
TRABAJO DE GRADO
Presentado como requisito parcial
Para optar al titulo de
BIOLOGO
PONTIFICIA UNIVERSIDAD JAVERIANA
FACULTAD DE CIENCIAS
CARRERA DE BIOLOGIA
Bogota, D.C
Junio, 2007

14
NOTA DE ADVERTENCIA
Articulo 23 de la Resolución No. 13 de Julio de 1946
�La Universidad no se hace responsable por los conceptos emitidos por sus alumnos
en sus trabajos de tesis. Solo velara por que no se publique nada contrario al dogma y
a la moral católica y por que las tesis no contengan ataques personales contra persona
alguna, antes bien se vea en ellas el anhelo de buscar la verdad y la justicia�

15
IDENTIFICACION Y LOCALIZACIÓN DE SECUENCIAS DE
REGULACIÓN DE LA EXPRESIÓN DE LA
FAMILIA DE GENES DE β-GLOBINA
EN DIFERENTES ESPECIES
NATALIA IVVON ACEVEDO LUNA
APROBADO
___________________________ __________________________
Leonardo Mariño Ramírez, Ph. D. Leonardo Lareo, Ph. D.
Director Asesor
____________________________ __________________________
Diana Alvarez, Ph.D. Carlos Manuel Estevez
Jurado Jurado

16
IDENTIFICACION Y LOCALIZACIÓN DE SECUENCIAS DE
REGULACIÓN DE LA EXPRESIÓN DE LA
FAMILIA DE GENES DE β-GLOBINA
EN DIFERENTES ESPECIES
NATALIA IVVON ACEVEDO LUNA
APROBADO
_______________________ _______________________
Ángela Umaña Muñoz Andrea Forero Ruiz
Decana Académica Directora Carrera de Biología
Facultad de Ciencias Básicas Facultad de Ciencias Básicas

17
AGRADECIMIENTOS
Al Dr. Leonardo Mariño Ramírez, PhD. Por la dirección científica y la acertada
orientación de este proyecto. Además por brindarme la oportunidad de realizar la
pasantía en el �National Center for Biotechnology Information� NCBI/NLM/NIH
Al Dr. Leonardo Lareo, PhD. Por su respaldo y sugerencias para el buen desempeñó
del proyecto.
Al Dr. David Landsman, PhD. por el soporte durante la realización del proyecto en el
�National Center for Biotechnology Information� NCBI/NLM/NIH
Al �National Center for Biotechnology Information�. NCBI/NLM/NIH Bethesda, MD.
Estados Unidos.

18
TABLA DE CONTENIDOS No.
1. INTRODUCCION
2. MARCO TEORICO Y REVISION DE LITERATURA
2.1. Regulación de la expresión génica
2.1.1. Secuencias cis de regulación
2.1.1.1. Promotores
2.1.1.1.1.Elementos Proximales de Regulación
2.1.1.2. �Enhancers�
2.1.1.3. Silenciadores
2.1.1.4. Aisladores
2.1.1.5. MARs/SASs
2.1.1.6. Elementos Aislantes
2.1.2. Factores de Transcripción
2.2. Cluster de genes de β-Globina
2.2.1. Evolución del cluster de los genes de β-Globina
2.2.2. Regulación de la expresión del cluster de genes de β-Globina
2.2.2.1. Locus de la región de control (LCR)
2.2.2.2. Regulación a través de modificaciones de la cromatina
2.2.2.3. Factores de transcripción propios de la expresión de los genes de β-
Globina
2.2.2.4. LCR y modificaciones de la cromatina
2.3. Evolución de los mecanismos de regulación del gen de β-Globina
3. FORMULACION DEL PROBLEMA Y JUSTIFICACION
3.1. Formulación del problema
3.2. Preguntas de la investigación
12
14
14
15
15
15
17
17
18
18
19
19
20
22
26
26
30
32
36
37
40
40
40

19
3.3. Justificación de la investigación
4. OBJETIVOS
4.1. Objetivo general
4.2. Objetivos específicos
5. MATERIALES Y MÉTODOS
5.1. Métodos para alcanzar el primer objetivo específico
5.1.1. Secuencias de regulación del locus de β-globina
5.2. Métodos para alcanzar el segundo objetivo específico
5.2.1. Obtención de secuencias para alinear
5.2.2. Determinación de las coordenadas en el cromosoma
5.3. Métodos para alcanzar el tercer objetivo específico
5.3.1. Ubicación de los genes de β-globina en cada especie
5.4. Métodos para alcanzar el cuarto objetivo específico
5.4.1. Secuencias de regulación conservadas
5.4.2. Secuencias en formato FASTA
5.5. Métodos para alcanzar el quinto objetivo especifico
5.5.1. Agrupación de secuencias según factor de transcripción
6. RESULTADOS Y DISCUSIÓN
6.1. Colección de sitios de regulación
6.1.1. Factores de Transcripción
6.2. Patrón de conservación de los sitios de regulación colectados
6.3. Localización de los genes del cluster de β-globina en las diferentes especies
6.4. Ubicación de los sitios de regulación en el genoma de cada especie
41
42
42
42
42
44
44
47
47
48
49
49
51
51
52
54
54
54
54
55
56
58
63

20
6.4.1. Conservación del orden en que se ubican los sitios de regulación
6.4.2. Locus de la Región de Control
6.4.3. Aisladores
7. CONCLUSION
8. RECOMENDACIONES
9. REFERENCIAS
10. ANEXOS
10.1 Anexo 1. Protocolo para determinar las coordenadas de las secuencias de
regulación conservadas en varias especies
10.2. Anexo 2: Protocolo para determinar las coordenadas de los genes de la
familia de β-globina en el genoma de cada especie incluida en el estudio
10.3. Anexo 3. Secuencias de regulación obtenidas a partir de la base de datos
TransFac®
10.4. Anexo 4. Sitios de regulación obtenidos a partir de la revisión
bibliográfica.
10.5. Anexo 5. Coordenadas de los sitios de regulación conservados en
diferentes especies
10.6. Anexo 6: Coordenadas de las secuencias colectadas según el factor de
transcripción que a estas se une
10.7. Anexo 7. Secuencias de regulación conservadas ordenadas de acuerdo a su
ubicación en el cluster de β-globina
10.8. Anexo 8. Secuencias en formato FASTA de los sitios de regulación
colectados.
72
76
79
81
84
85
92
92
98
103
105
107
110
113
117

21
RESUMEN
La expresión de los genes de β-globina es específica a un tipo de tejido celular y a la
etapa del desarrollo. El mecanismo que controla la especificidad de la expresión de
cada gen del cluster requiere de elementos de regulación proximales y dístales, como
lo es el Locus de la Región de Control (LCR). A pesar de la gran cantidad de
información disponible acerca de la regulación de los genes de β-globina, aun hay
mucho por entender acerca del mecanismo que regula tal patrón de expresión. El
objetivo de este proyecto es determinar, a través de métodos computacionales, que tan
similares son los elementos de regulación del cluster de β-globina en diferentes
vertebrados. Se colectaron a través de la base de datos TransFac® y de revisión crítica
de la literatura, 122 secuencias de regulación del cluster de β-globina en diferentes
especies. Basándose en alineamientos de secuencias, se determinó el número de
secuencias posiblemente conservadas en humano, chimpancé, ratón, rata, perro y
pollo, así como las coordenadas para los sitios de regulación presentes en cada
especie. En el cluster de β-globina de los mamíferos se ubicaron los sitios
hipersensitivos HS2, HS3 y HS4, y en el del ave los sitios HS2 y HS4, los cuales no
muestran homología con los encontrados en los mamíferos. De forma similar, se
mapearon los genes de β-globina logrando así comparar la organización del cluster en
los mamíferos y el ave. Los datos colectados en este proyecto aportan la materia prima
para el estudio del patrón de evolución de los mecanismos de regulación de la
expresión de los genes de β-globina.

22
ABSTRACT
The β-globin genes are expressed in a tissue and stage specific manner. The
mechanism controlling the stage specific expression of each gene of the cluster
requires both, proximal and distal elements as the Locus Control Region (LCR).
Despite the vast amount of information regarding the β-globin genes regulation, the
actual mechanism of regulation remains still unclear. This project aims to determine
how similar the β-globin gene regulatory elements are among some vertebrates. 122
transcription factor binding sequences from the β-globin cluster of different species
were collected using TransFac® data base and also from a bibliographic review. The
number of conserved sequences among human, chimp, mouse, rat, dog and chicken
were determined based on sequence alignments. The chromosome coordinates of each
regulatory site as well as of each gene were determined to locate them in the cluster on
each species under study. The location of the sites and genes enabled to establish a
comparison of their arrangement within the β-globin cluster. It was possible to
recognize the mammal conserved sites HS2, HS3, HS4 which are part of the LCR. On
the other hand, the HS2 and HS4 were located in the chicken cluster, but these sites
were not homologous to those of mammals. The comparison of the collected
sequences made possible to find a region in the mammals� cluster which may be
homologous to an enhancer described in chicken. The data collected in this project
represents the working material to establish the pattern of evolution of the β-globin
cluster regulatory mechanisms.

23
1. INTRODUCCION
El advenimiento de proyectos de secuenciación de genomas completos ha hecho
posible conocer la secuencia de varios genomas eucariotas entre otros, S. cervisiae,
1997; C. elegans, 1999; D. melanogaster, 2000; humano, 2001; ratón, 2002; pollo,
2004 (el año hace referencia a la publicación del primer borrador de la secuencia). Lo
cual representa una gran cantidad de información de la cual aun hay mucho por
interpretar. Es por esto que el siguiente reto es predecir la función de cada uno de los
genes del genoma. Un acercamiento a dicho entendimiento se logra a través del
estudio de los mecanismos de regulación y cómo estos se han conservado a través del
tiempo. La realización de este proyecto aporta un acercamiento a la obtención de ese
conocimiento
Los mecanismos que gobiernan la regulación de la expresión específica, a un tipo
celular y a una etapa del desarrollo, del gen de la β-globina han sido ampliamente
estudiados, muchos de estos estudios han caracterizado varias secuencias de
regulación y los respectivos factores de transcripción que se unen a estas (Hardison, et
al, 1998; Hardison, 2001; King, et al, 2005; Dorschner, et al. 2004). Sin embargo, a
pesar de todo el conocimiento que hay acerca de la regulación transcripcional de la
expresión de los genes que codifican para β-globina, poco se conoce acerca de la
dinámica evolutiva que dio origen a esos mecanismos de regulación.
Este proyecto propone el primer paso en el estudio de la evolución del mecanismo de
regulación propio a la expresión de los genes de β-globina, mediante la colección y
ubicación de secuencias de regulación en este cluster en diferentes especies.
A través de la búsqueda en la base de datos TransFac® y en la literatura, se colectaron
las secuencias de unión a factores de transcripción dentro del cluster de β-globina y se
identificaron los posibles sitios conservados en las diferentes especies incluidas en el

24
estudio (humano, chimpancé, ratón, rata, perro y pollo). Cada secuencia de regulación
conservada se ubico en el genoma de cada especie y así fue posible establecer
comparación de la organización del cluster en las diferentes especies. Así mismo, con
base en la ubicación de las secuencias de regulación se reconocieron regiones en el
cluster donde pueden localizarse más sitios de regulación aun no mapeados.
El reconocimiento de elementos de regulación dentro de las regiones de DNA no
codificante representa un reto por que se trata de secuencias muy cortas que se pueden
repetir al azar en el genoma y distinguir cuales de esas son funcionales en términos de
regulación, es una predicción cuyos criterios son difíciles de limitar (Mariño-Ramírez,
et al. 2004). Este proyecto, mediante la colección y ubicación de secuencias aporta las
bases para la posterior búsqueda de más elementos de regulación.
Así, los datos colectados en este proyecto permiten un acercamiento al escenario
general de la regulación del cluster de β-globina en diferentes especies al generar un
mapa con la ubicación de las secuencias de regulación respecto a la de los genes en el
cluster de cada especie, permitiendo reconocer las regiones en el cluster propensas a
contener elementos de regulación, así como patrones conservados en la organización
de las secuencias en el cluster.
De esta forma, se aporta la información que facilitará el uso de algoritmos
computacionales para la búsqueda de elementos de regulación así como para
determinar los motivos de regulación conservados a partir de las secuencias
colectadas. Siendo entonces la base a partir de la cual se puede estudiar el patrón de
evolución de los mecanismos de regulación de los genes de β-globina.

25
2. MARCO TEORICO Y REVISION DE LITERATURA
2.1. Regulación de la expresión génica
Todas las células requieren un programa especial de regulación de la expresión de sus
genes. Programa que le permite a la célula tener unos genes activos mientras otros
están silenciados según los requerimientos en un determinado momento.
La iniciación de la trascripción es el paso más importante en la regulación de la
expresión de un gen. El control de la trascripción es un mecanismo donde participan
secuencias de acción cis y factores de transcripción. Las secuencias de acción cis
generalmente están en la región 5� del sitio de inicio de la trascripción aunque algunas
están corriente abajo o incluso dentro de la secuencia del gen (Levine y Tjian, 2003).
Estas secuencias son la región de reconocimiento para los factores de acción trans los
cuales se unen a las secuencias cis generando en el DNA modificaciones que pueden
activar o reprimir la transcripción de determinado gen. Generalmente, los mecanismos
de regulación de la expresión de un gen involucran varias secuencias cis y por tanto
varios factores de trascripción, la interacción entre dichos factores y de estos con las
secuencias cis hacen del proceso de regulación un complejo con múltiples opciones de
represión y activación (McClean, 1997).
En el proceso de regulación de la expresión de un gen interviene la expresión de otros
genes y sus secuencias cis y factores trans haciendo parte de una cascada de eventos
genéticos, dado que los factores de trascripción en si mismos también son objeto de
regulación (Struhl, 2001).

26
2.1.1. Secuencias cis de regulación
2.1.1.1.Promotores
Los promotores son secuencias ubicadas en el lado 5� del sitio de inicio de la
transcripción, la función de estas secuencias es indicar donde esta el sitio a partir del
cual se inicia la transcripción. Algunas de las secuencias de promotores se han
conservado y son iguales en muchos genes, estas secuencias conservadas como la caja
TATA, el elemento BRE y el sitio INI hacen parte del promotor basal (Blackwood y
Kadonaga, 1998).
Corriente arriba al promotor basal, entre -50 a -200pb del sitio de inicio de la
transcripción, se localizan el promotor proximal el cual contiene varias secuencias a
las que se unen un subgrupo de factores de transcripción: SP1, CTF y CBF. Por otra
parte, la caja CAAT se ubica entre 50 a 130pb del sitio de inicio (McClean, 1997). A
la caja CAAT se une el factor de trascripción C/EBP del ingles �CAAT Box Enhancer
Binding Protein�.
En la combinación apropiada, los promotores pueden dirigir a la RNA polimerasa II
par el inicio de la transcripción.
2.1.1.1.1. Elementos Proximales de Regulación
Los elementos proximales hacen parte del promotor basal y son requeridos para la
correcta regulación espacio-temporal de un gen. Dentro de los elementos de
regulación proximal se incluyen la caja TATA, el sitio de reconocimiento de TFIID
(BRE), el iniciador (INR) y el promotor corriente abajo (DPE) (Wray, et al. 2003).
La caja TATA es una región rica en adeninas y timinas que se localiza a más o menos
a partir del nucleótido 30 corriente arriba del sitio de inicio de la transcripción. Este

27
sitio es reconocido por la subunidad TBP (�TATA binding protein�) del complejo
TFIID. Inmediatamente corriente arriba de la caja TATA se encuentra el sitio BRE el
cual incrementa la afinidad de la proteína TFIID a la caja TATA al contener otro sitio
de reconocimiento para este complejo. El sitio iniciador INR es una secuencia
conservada que contiene el sitio de inicio de la transcripción, su función es dirigir la
iniciación de la transcripción. Finalmente el elemento DRE, se localiza a cerca de
30pb corriente abajo del sitio de inicio y es reconocido por el factor asociado a TBP
(Butler y Kadonaga, 2001; Levine y Tjian, 2003) (Figura 1)
Además del promotor basal y el proximal, la mayoría de genes contienen sitios de
unión proximales localizados en el extremo 5� del promotor. Los factores que se unen
a estos sitios pueden servir como elementos que atraen los �enhancers� dístales a los
promotores (Calhoun, et al. 2002).
Figura 1. Estructura general de un gen en un mamífero con la ubicación aproximada
de los sitios de regulación cis. (Tomada de Levine y Tjian, 2003)

28
2.1.1.2. �Enhancers�
Los organismos eucariotas son capaces de expresar sus genes en respuesta a
condiciones ambientales y del desarrollo, mostrando patrones de expresión génica
complejos, diversos y a la vez precisos. Lo anterior sucede gracias a los �enhancers�,
secuencias de DNA relativamente cortas que contienen múltiples sitios de unión a
proteínas que activan la transcripción.
Así, los �enhancers� son secuencias que incrementan la tasa de transcripción de un
gen. Las regiones de control de la transcripción generalmente contienen varios
�enhancers� que pueden variar en tamaño desde 50pb hasta 1.5kb (Blackwood y
Kadonaga, 1998). Cada una de esas secuencias desarrolla una función específica
como la activación de un gen en un tipo celular determinado y en un estado particular
del desarrollo. Un gen, puede tener entonces varias de estas secuencias �enhancer�,
cada una de las cuales contribuye de forma tanto espacial como temporal a la
regulación de la expresión del gen.
Este tipo de secuencias de acción cis pueden localizarse tanto corriente arriba como
corriente abajo del gen y pueden actuar desde distancias tan amplias como ~50kb.
Debido a que actúan a grandes distancias del gen, generalmente requieren que el DNA
adquiera conformaciones en bucles (�loops�) lo cual acerca la proteína activadora,
unida al �enhancer� distal, a los complejos de proteínas asociados con el promotor.
Los �enhancers� generalmente activan la transcripción de forma específica al tejido,
es decir que inducen la transcripción de un gen en uno o algunos tipos celulares
(Blackwood y Kadonaga, 1998).

29
2.1.1.3. Silenciadores
A los silenciadores se unen complejos de proteínas represoras interfiriendo con la
actividad del promotor de manera que reducen la expresión del gen. Los silenciadores
tienen una función antagonista con respecto a los �enhancers� y al igual que estos, los
silenciadores pueden actuar desde grandes distancias (Harju, et al, 2002).
2.1.1.4. Aisladores
Los aisladores son secuencias de DNA con sus respectivas proteínas de unión cuyo
rol es establecer o mantener los límites entre dominios de cromatina transcripcional.
Estas secuencias tienen un tamaño aproximado de 0.5 a 3kpb (Blackwood y
Kadonaga, 1998) y protegen el locus de la heterocromatina que lo rodea. Pueden
actuar como elementos que flanquean el dominio transcripcionalmente activo y
también pueden evitar la acetilación de las histonas (Harju, et al, 2002) haciendo más
difícil la unión de los factores de trascripción al DNA.
Existen dos tipos de aisladores, los que establecen dominios que separan promotores
de �enhancers� impidiendo su interacción (�enhancer-blocking�), y los que generan
una barrera que evita la extensión de la heterocromatina (�barrier insulators�). Los
aisladores de bloqueo de �enhancers� también protegen contra algunos tipos de
represores transcripcionales. Algunos aisladores pueden tener las dos funciones:
bloquear la actividad de los �enhancers� y generar barreras (West, et al. 2002;
Gaszner y Felsenfeld, 2006).
2.1.1.5.MARs/SASs
Estas son regiones de unión a matrices o regiones de unión a �scaffold�, promueven la
unión a la matriz nuclear generando la formación de bucles de DNA, la formación de
estos bucles puede alejar el locus de los efectos negativos de la cromatina alrededor.

30
Así mismo, la unión a matrices puede facilitar la activación de la trascripción con la
formación de los �loops� de DNA acercando los elementos cis de regulación. De este
modo, los elementos MARs pueden ayudar en la interacción de secuencias cis de
regulación distantes y promotores (Harju, et al, 2002).
2.1.1.6. Elementos Aislantes
Estos elementos se pueden ubicar en diferentes posiciones dentro del locus y pueden
tener un rol restrictivo de la transcripción cuando se asocian a una proteína de unión al
DNA. Es posible que los elementos aislantes estén relacionados con mantener
constante el estado abierto o cerrado de la cromatina. Así mismo, también contienen
elementos de acción cis que previenen la acción negativa de cromatina externa al
dominio.
Los elementos aislantes se pueden ubicar tanto corriente arriba como corriente abajo
del locus de la β-globina, mas aun se pueden localizar dentro del locus, demarcando
la expresión regulada por una determinada etapa del desarrollo en un subdominio
(Harju, et al, 2002).
2.1.2. Factores de Transcripción
Los factores de transcripción son proteínas que se unen a las secuencias cis de
regulación de manera especifica. Estas proteínas funcionalmente tienen dos dominios
uno de los cuales es requerido para la unión al DNA y el otro para la activación de la
transcripción (Wray, et al. 2003).
Un gen puede estar regulado por varias secuencias de acción cis y por tanto ser objeto
de la influencia de varios factores de transcripción cuyos sitios de unión están
repartidos a lo largo del cromosoma. Algunos factores pueden estar restringidos a un
tipo celular determinado y operar únicamente durante etapas específicas del desarrollo.

31
Por ejemplo GATA-1 es un factor de transcripción específico a los eritrocitos. GATA-
1 puede actuar como represor o activador dependiendo del contexto de su secuencia de
unión y de la interacción con otras proteínas. GATA-1 actúa como activador cuando
se une al promotor del gen γ-globina o como represor cuando se une al silenciador del
gen ε-globina. De esta forma, GATA-1 es requerido para determinar que gen se
expresa durante la maduración de los glóbulos rojos (Harju, et al. 2002; Welch, et al.
2004).
La transcripción eficiente requiere la combinación y la acción en sinergia de múltiples
factores, varias combinaciones diferentes de factores son capaces de activar la
transcripción. Como consecuencia puede haber un limitado número de factores de
transcripción que pueden organizarse en diferentes combinaciones cada una de las
cuales representa una forma biológicamente distinta de regular la expresión de un gen
(Struhl, 2001).
La capacidad de los factores de transcripción de unirse a una secuencia específica de
DNA la confiere el dominio de unión el cual hace parte del motivo estructural del
factor. Entre los motivos estructurales mas comunes están el homeodominio, �helix-
loop-helix� (HLH), �Zinc fingers�, �Leucine zipper� y �Winged Helix� (McClean,
1997). En general, una hélice alfa en el dominio de unión de un factor de
transcripción, interactúa con el surco mayor del DNA en la secuencia de unión. Los
factores de transcripción son proteínas que suelen tener un solo dominio de unión al
DNA y uno o más dominios de activación o represión a los cuales se podrían unir
otras proteínas (Lemon y Tjian, 2000).
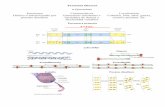
2.2. Cluster de genes de β-Globina
En general en las aves y mamíferos, los genes que codifican las globinas están
agrupados en dos clusters en uno de los cuales se ubican los genes para α-globina y en
otro, en un cromosoma diferente, los de la β-globina.

32
En los humanos, el cluster de los genes de la β-globina consiste en una familia de
cinco genes funcionales, un pseudo gen y un elemento distal de regulación ubicado
corriente arriba. Los cinco genes tienen un patrón de expresión específico al tejido y al
estado del desarrollo y se ubican espacialmente en el cromosoma 11 (70kb) en el
mismo orden en que se expresan durante ontogenia (Figura 2) (Harju, et al, 2002).
Figura 2. A. Estructura del cluster de genes de β-globina en humano. El locus consiste
en cinco genes funcionales y un pseudo gen. El orden de los genes en el cromosoma
coincide con el orden de su expresión durante el desarrollo. Corriente arriba a los
genes se encuentra el locus de la región de control, LCR, caracterizado por cinco sitios
hipersensibles. B. Patrón de expresión de los genes de β-globina. El eje �x� es la edad
del feto en semanas y el eje �y� la expresión del gen en porcentaje del total de globina
expresada. (Figura tomada de Harju, et al. 2002).

33
El gen ε-globina se expresa en el saco embrionario donde ocurre la eritropoyesis
embrionaria o primitiva durante las seis primeras semanas de gestación en los
eritrocitos primitivos, los cuales son nucleados. Mientras el gen ε-globina se expresa,
los genes γ- y β-globina están silenciados. Así mismo, cuando la expresión de los
genes Gγ- Aγ-globina se activa en las células hematopoyeticas definitivas en el hígado
del feto, el gen ε-globina es silenciado. Finalmente, el gen β-globina, y el δ-globina,
se activan en la medula ósea y en el bazo, donde ocurre la eritropoyesis adulta
definitiva. Recíprocamente, al activarse β- y δ-globina, los genes γ-globina no se
expresan. En el adulto δ-globina se expresa en bajos niveles mientras que β-globina es
producida en abundancia (Harju, et al, 2002).
2.2.1. Evolución del cluster de los genes de β-Globina
Pocas proteínas han sido estudiadas en tantos organismos como lo han sido las
diferentes formas de globina (Hardison, 2001; Hardison y Miller, 1993; Wheeler, et al.
2001). La familia de las globinas ha sido estudiada fisiológicamente en bacterias,
hongos, plantas y animales y su rol varia desde catalizar la combinación de oxigeno y
oxido nítrico para formar nitrato en bacterias, levaduras e invertebrados, hasta el
transporte de oxigeno en la sangre en los vertebrados (Hardison, 1998). Esta diferencia
de funciones ilustra la adquisición de nuevos roles por parte de genes estructurales
preexistentes lo cual requiere cambios tanto en las secuencias que codifican como en
los elementos de regulación de los genes (Hardison, 1998).
La comparación de los genes de la β-globina en varias especies de mamíferos
placentarios ha permitido estimar que en la evolución de este cluster han ocurrido
eventos de duplicación de los genes a partir de un ancestro común hace
aproximadamente 450 millones de años. Según Wheeler y colaboradores (2001), estos
eventos de duplicación permitieron la evolución de las diferentes formas de globina de
acuerdo a los requerimientos de transporte de oxigeno en las diferentes etapas del

34
εI εII ψβX βC εIII εIV ψβZ βA εV εVI ψβY βF
ε γ ψη δ β
ε Gγ Aγ ψη δ β
ε γ ψδ β
y bh0 bh1 bh2 bh3 b1 b2
0 140KB
desarrollo. Se ha postulado que dicho gen ancestral tenía 3 exones separados por dos
intrones (Hardison y Miller, 1993).
El cluster de β-globina incluye los genes ε, γ, ψ, δ y β sin embargo, las duplicaciones
en el cluster ocurrieron de manera independiente en los linajes de las aves y los
mamíferos. El proceso de duplicación y divergencia en los mamíferos ha generado los
cluster que se muestran en la figura 3 donde se ilustra la organización de los genes de
la β-globina en diferentes mamíferos. La distancia entre los genes es variable y en
muchos casos es resultado de la inserción de retrotransposones (Hardison y Miller,
1993).
Cabra (Capra hircus) Galago (Galago alleni) Humano (Homo sapiens) Conejo (Oryctolagus cuniculus) Ratón (Mus musculus)
Figura 3. Representación de los genes de β globina en diferentes mamíferos
orientación 5� a 3�. Las distancias son aproximadas. (Tomado de Hardison y Miller,
1993).

35
En las aves, el cluster de la β-globina tiene una organización diferente a la del cluster
en los mamíferos placentarios teniendo dos genes embrionarios en los extremos del
locus y que codifican para ρ-globina y ε-globina, y dos genes para β-globina uno de
los cuales codifica para βH-globina y se expresa cuando el ave sale del huevo y otro
βA-globina se expresa en los glóbulos rojos del ave adulta (Hardison, 2001).
Según afirma Hardison, (2001), los clusters de la β-globina de estos dos taxa, aves y
mamíferos placentarios, se consideraban ortólogos debido a que en las aves es ρ-β-ε y
en los mamíferos placentarios es ε-γ-δ-β , por lo que se pensaba que provenían del
mismo cluster en el ultimo ancestro común entre las aves y los mamíferos. Sin
embargo, el descubrimiento del gen ω-globina en los marsupiales, hizo reconsiderar
esa idea orientándola a que el gen de los Eutheria ε-γ-δ-β sea descendiente de un
cluster diferente al que dio origen al cluster ρ-β-ε- globina de las aves.
El gen ω-globina, presente en los marsupiales, se expresa después del nacimiento.
Holland y colaboradores (1997) realizaron un estudio parcial de la secuencia de
aminoácidos en ω-globina indicando que esta proteína esta mas relacionada con la β-
globina de las aves que con la de los mamíferos placentarios y marsupiales.
Estudios mas recientes indican que el gen ω-globina forma un grupo hermano con los
genes ρ-β-ε-globina de las aves, al estar mas relacionado con los de este cluster en las
aves que con cualquier gen β-globina en mamíferos incluyendo los del marsupial. Así,
este grupo esta claramente separado de la β-globina en Eutheria y marsupiales.
Además, estos estudios muestran que el gen ω-globina esta ubicado en un cluster
diferente al de la β-globina (Wheeler, et al. 2001).
De esta forma los marsupiales tienen un gen ω-globina similar a los genes de β-
globina en las aves y además tienen el cluster de gen β-globina el cual es similar al de
los mamíferos placentarios. Es posible que ambos clusters (ω-globina y β-globina)

36
hayan sido heredados de un ancestro común con las aves y los mamíferos. De haber
sido así, los genes de la β-globina de los mamíferos placentarios y las aves no serian
ortólogos, es decir no habrían evolucionado directamente a partir de un mismo gen
ancestral. Según esto, el cluster ancestral se debió duplicar antes de la divergencia de
aves y mamíferos y una ves duplicados uno de los cluster divergió para dar origen al
cluster de la β-globina ε-γ-δ-β- encontrado en los mamíferos placentarios y el cluster
ε-β de los marsupiales. Mientras que, la otra duplicación del cluster ancestral bebió dar
lugar al cluster ρ-β-ε de las aves, y al gen ω-globina de los marsupiales (Figura 4).
Figura 4. Modelo de evolución independiente de los cluster de β-globina en aves y
mamíferos. El diagrama muestra el cluster ancestral con los genes pβ (proto globina).
El modelo propone la duplicación del cluster ancestral para generar el ancestro de los
genes β-globina de los eutheria (eβ1 y eβ2) y el ancestro de los genes β-globina de las
aves (aβ1 y aβ2). La vía de evolución relacionada con los genes β-globina de
mamíferos placentarios se muestra en rojo y la relacionada con los genes β-globina de
las aves en azul. La duplicación de los genes esta indicada con círculos amarillos y la
separación de los genes por eventos de especiación esta señalada con rombos (Figura
tomada de Hardison, 2001).

37
2.2.2. Regulación de la expresión del cluster de genes de β-Globina
La transcripción del complejo de genes β-globina es regulada por elementos
proximales y por un elemento distal conocido como el locus de la región de control,
LCR �Locus Control Region� además de las secuencias de acción cis antes enunciadas
y que son típicas de la regulación de expresión de un gen. La forma como es regulada
la expresión del gen involucra interacción de varias moléculas entre si y con sus sitios
de unión al DNA. Además, las modificaciones en la cromatina también puede alterar
el patrón de expresión del gen.
2.2.2.1. Locus de la región de control (LCR)
El locus de la región de control físicamente esta definido por la presencia de cinco
sitios hipersensibles (HS) a DNasa I localizados entre 2 y 22kb corriente arriba del
gen de la ε-globina (Figura 2) los sitios HS son susceptibles a digestión con DNasa I
debido a que en ellos hay disrupción del nucleosoma, esto permite que el DNA este
accesible para la trascripción y los factores de remodelación de la cromatina.
Contrario a la mayoría de las secuencias de regulación cis, LCR funciona a largas
distancias para activar la expresión del gen y mantener abierto el dominio de
cromatina del gen β-globina en los eritrocitos. (Harju, et al, 2002; Kim y Dean, 2004)
En la tabla 1. Se describe la actividad de cada sitio HS dentro de LCR.
Los primeros estudios en la regulación del locus de la β-globina sugieren una vía de
activación transcripcional que incluye la pre-activación del locus la cual es detectada
como un incremento en la susceptibilidad a la digestión con DNasa I. Una vez
preactivado el locus, los genes pueden ser activados. Este modelo que involucra
competencia transcripcional antes de la activación de la trascripción es reafirmado por
la acción de la región LCR en la regulación del locus de la β-globina. Puesto que LCR
actúa primero en el estado del desarrollo previo a la activación de los genes para ser
transcritos, dejando a los cinco genes del locus competentes pero transcripcionalmente

38
inactivos. Una vez en dicho estado, cada gen es regulado por �enhancers� y represores
específicos a cada estado del desarrollo (Blackwood y Kadonaga, 1998).
Tabla 1. Actividad de cada región hipersensible dentro de LCR (Actividad observada
en ratones transgenicos) (Tomada de Harju y Colaboradores, 2002)
Sitio hipersensible Actividad
5�HS4
Activación de la trascripción
Expresión independiente de la posición
Expresión independiente del numero de copias
5�HS3
Activación de la trascripción
Expresión independiente de la posición cuando esta ligado al gen de la
β-globina
Expresión dependiente del numero de copias cuando esta ligado al gen
de la β-globina
Expresión dependiente de la posición cuando esta ligado al gen Aγ-
globina
Actividad de remodelación de la cromatina.
5�HS2
Actividad de �enhancer�
Expresión independiente de la posición.
Expresión dependiente del número de copias.
Tiene secuencias de caja E (sitios de unión para factores de
trascripción de dominio �Helix-loop-helix�)
Posee secuencias de unión a: GATA-1, NF-E2, Sp1, Ap-1, USF; todas
son necesarias para la actividad de �enhancer�.
La deleción de un sitio de unión a un factor de trascripción no impide
la expresión independiente de la posición.
No tiene actividad de �enhancer� por si mismo, requiere la presencia
de otros sitios LCR HS
5�HS1 No tiene actividad de activación de la trascripción
5�HS5 Tiene secuencias homologas a MARs y puede funcionar como
aislador.

39
Estudios en el locus de β-globina en pollo han mostrado que el establecimiento del
estado competente se relaciona con un incremento de la acetilación de las histonas y
en la susceptibilidad a la digestión con DNasa I, eventos que están relacionados pues
la hiperacetilación de las histonas conlleva al desdoblamiento del DNA que a su vez
facilita la accesibilidad de factores al DNA (Factores como DNasa I o factores de
transcripción) (Blackwood y Kadonaga, 1998).
La función de la región LCR fue definida con base a estudios con transgenes. A partir
de dichos estudios se observo que los sitios HS actúan en sinergia para interactuar
directa o indirectamente con los elementos de regulación proximales al gen ejerciendo
la regulación apropiada. Así mismo se pudo observar que la función de LCR como
unidad puede depender de la orientación debido a que 5�HS5 tiene función de aislador,
si la secuencia LCR es invertida estaría aislando los genes del cluster de la interacción
con LCR y por tanto estaría silenciándolos (Harju, et al, 2002).
Choi y Engel, (1988) propusieron la hipótesis de competencia para explicar la forma
como el sitio LCR interactúa con los genes de la β-globina de acuerdo a la etapa del
desarrollo en aves. Dicha hipótesis afirma que los promotores de cada gen en el cluster
compiten por la interacción con el sitio LCR durante el desarrollo de forma que,
durante el estado embrionaria la disponibilidad de factores específicos a este estado
favorecen la interacción entre LCR y el gen ε-globina; mientras que en el estado
adulto, la presencia de factores específicos de la edad adulta favorecen la interacción
entre LCR y el gen β-globina. Además de la competencia entre los promotores para el
cambio de gen que se expresa en determinado momento del desarrollo, en el caso de
los mamíferos, el orden normal de los genes en el locus de β-globina también
desempeña un rol importante en la temporalidad de la expresión de los genes (Enver,
et al. 1990). Así, el contexto del locus (orden de los genes en el cluster) y los factores
de transcripción que influyen en la interacción entre LCR y los genes de β-globina
actúan en sinergia para regular la expresión de los genes durante el desarrollo
(Tanimoto, et al. 1999; Ryan, et al. 2000).

40
De otra forma, en el promotor distal del gen ε-globina hay un silenciador el cual
controla la represión autónoma de la expresión del gen ε-globina durante el estadio
fetal y la vida adulta. Algunos estudios mutacionales en ratones sugieren que este
silenciador contiene secuencias importantes para la activación del transgen γ-globina
durante el estado embrionario del ratón. Otros elementos asociados a este silenciador
son repeticiones directas (DR) proximales al promotor ε-globina a las cuales se unen
proteínas eritroides definitivas (DRED) esta unión interfiere con un factor de unión al
promotor llamado �Kruppel-like� factor (EKLF) De esta forma, el silenciador detiene
la expresión del gen ε-globina (Harju, et al, 2002).
Es claro que LCR tiene un rol en la expresión de los genes de β-globina sin embargo,
aun no es claro el mecanismo por el cual el sitio LCR interactúa con los genes de la
globina Harju y colaboradores (2002) así como Li y colaboradores (2002) enuncian
cuatro posibles formas de interacción, estas son:
• Modelo �Loop�
Este modelo sugiere que las regiones 5�HS se doblan formando un complejo en cuyo
centro están los sitios HS activos a los cuales se unen los factores de transcripción,
además también están las secuencias flanqueantes limitando dicho complejo. Esta
estructura se acerca al promotor proximal, mediante un loop en el DNA, llevando
consigo los factores de trascripción de modo que puedan interactuar con el complejo
de trascripción unido al promotor activando así la expresión del gen.
• Modelo �Tracking�
Este modelo propone que los factores y co-factores de transcripción se unen a las
secuencias LCR formando un complejo de activación que migra linealmente sobre el
DNA hasta encontrarse con la maquinaria de transcripción sobre el promotor,
activando así la transcripción.

41
• Modelo de migración facilitada
Este modelo combina aspectos del modelo �loop� y del modelo �tracking�. Los
factores de transcripción se unen a las secuencias 5�HS del LCR y este complejo
migra a lo largo de la cromatina hasta que encuentra el promotor momento en el que
se forma un �loop� estable para asegurar la interacción entre el promotor y las
proteínas unidas a los sitios 5�HS.
• Modelo de enlace
Sugiere que hay unión de factores de manera secuencial con proteínas facilitadotas a
través del locus las cuales ayudan a definir el dominio transcipcional. Los factores de
transcripción unidos a los promotores y los sitios HS se ponen en contacto a través de
una cadena de factores facilitadotes, los cuales no se unen al DNA.
2.2.2.2. Regulación de la expresión del gen a través de modificaciones de la
cromatina
Las modificaciones en la cromatina tienen un rol importante en el control de la
transcripción (Mariño-Ramírez, et al, 2005). Los cambios generales de la cromatina
como acetilación, fosforilación y metilación tienen un efecto en la activación de un
locus:
• Acetilación
La acetilación ocurre en las histonas y varía de acuerdo a la fase del ciclo celular. Al
ocurrir en las histonas, la acetilación genera disrupción del contacto del DNA con el
nucleosoma permitiendo a los factores de transcripción acceder al DNA y activar la
expresión de un gen. De esta forma, la hiperacetilación esta asociada con la activación
de la transcripción de un locus. Los factores que influyen en la acetilación de la

42
cromatina del locus de la β -globina pueden ser puntos importantes de control de la
expresión del gen. Por ejemplo, La acetilación en la histona H4 induce la formación
del complejo de preiniciación de la transcripción con el factor TFIID, el factor de
trascripción NF-E2, propio de los eritrocitos, se unen a TFIID haciendo posible la
activación del gen. De esta forma, la acetilación puede servir como un indicador de la
actividad transcripcional en el locus del gen de la β �globina (Harju, et al. 2002).
• Fosforilación
El efecto de la fosforilación de la cromatina en la regulación de la expresión de un gen
es similar al de la acetilación. La fosforilación de la histona H3 interrumpe la
interacción DNA-nucleosoma incrementando la accesibilidad de factores de
transcripción al DNA. Las vías PAPkinasa y p38, las cuales son inducidas en respuesta
a estrés, activan la fosforilación de H3 (Harju, et al. 2002).
• CpG Metilación
Contrario al efecto en la iniciación de la trascripción que tienen la acetilación y la
fosforilación de la cromatina, la metilación actúa como una barrera a la formación del
complejo de preiniciación de la transcripción y a la accesibilidad de los factores de
transcripción al DNA. La metilación no afecta la conformación del nucleosoma. Las
áreas activas de la cromatina generalmente no están metiladas y la metilación de las
islas CpG en regiones de promotores están asociadas con pérdida de la
hipersuceptibilidad a DNasa-I (Harju, et al. 2002).
• Complejos de remodelación de la cromatina
SWI/SNF: Están relacionados con regulación de la estructura de la cromatina y
transcripción mediante la movilización de nucleosomas por el rompimiento y
reestablecimiento de contactos entre el DNA y las histonas.

43
PYR: Este complejo se une a secuencias de DNA ricas en pirimidinas entre los genes
y- y δ-globinas. La unión depende tanto de la secuencia como del tamaño de esta. El
complejo PYR se puede unir a los elementos aislantes de los genes ε-γ-δ-β-globina
influyendo en el cambio de la estructura de la cromatina en el locus durante el cambio
de γ a β-globina.
HATs: Histona acetil transferasas. Transfieren grupos acetil a lisinas específicas en
las proteínas reduciendo la carga positiva en ellas por tanto disminuyendo la unión de
estas proteínas al DNA, cargado negativamente. Sin embargo, la acetilación del factor
GATA-1 cambia la conformación de la proteína e incrementa su capacidad de unión al
DNA (Harju, et al. 2002).
2.2.2.3. Factores de transcripción propios de la expresión de los genes de β-
Globina
Se han descrito y caracterizado varios factores de trascripción que controlan la
expresión del gen de la β -globina. Estos factores forman una red de interacciones
proteína-proteína, proteína-DNA, con los promotores, con las regiones HSs de LCR y
con otras regiones ínter génicas de acción �cis. En la tabla 2 se recopila dicha
información con base a la revisión de Harju y colaboradores (2002).

44
Tab
la 2
. Fa
ctor
es d
e tra
nscr
ipci
ón q
ue in
terv
iene
n en
la re
gula
ción
de
la e
xpre
sión
de lo
s
gene
s de β-
glo
bina

45
Fact
or
Dom
inio
Si
tios d
e un
ión
Mec
anis
mo
Func
ión
NF-
E2
Car
acte
rizad
o en
hu
man
o (T
albo
t, et
al.
1990
; N
ey, e
t al.
1990
) y
en
rató
n (C
han,
et
al. 1
993)
Het
erod
imer
o
Leuc
ine
zipp
er
Tien
e do
s su
buni
dade
s m
af
p45
y p1
8.
Las s
ubun
idad
es s
e un
en c
omo
hom
odim
eros
o
hete
rodi
mer
os a
M
AR
Es q
ue so
n sit
ios
de re
cono
cim
ient
o pa
ra
prot
eína
s maf
El p
oten
cial
de
activ
ació
n de
l com
plej
o N
F-E2
es
mod
ulad
o po
r la
inte
racc
ión
de la
su
buni
dad
p45
con
dife
rent
es su
buni
dade
s m
af
lo q
ue re
sulta
en
dife
rent
e es
peci
ficid
ad d
e la
re
gula
ción
.
La u
nión
de
NF-
E2 a
sitio
s M
AR
E de
ntro
de
LCR
del
ge
n de
la β
-glo
bina
es
nece
saria
par
a la
act
ivac
ión
de la
tras
crip
ción
y p
ara
la
form
ació
n de
sitio
s HSs
de
ntro
de
LCR
.
GA
TA
-1
Des
crito
en
hum
ano
(Mer
ika
y O
rkin
, 19
93)
GA
TA z
inc-
finge
r que
se
cara
cter
iza
por
pode
rse
unir
a la
re
gión
con
sens
o W
GA
TAR
Se u
ne a
los p
rom
otor
es
de lo
s ge
nes d
e la
β
glob
ina
y a
los s
itios
5�
HS1
al 5
.
Func
iona
com
o ac
tivad
or o
re
pres
or d
epen
dien
do d
el
cont
exto
de
la se
cuen
cia
de
unió
n y
de su
s in
tera
ccio
nes c
on o
tras
prot
eína
s.
Inte
ract
úa c
on o
tros
fact
ores
de
trans
crip
ción
co
mo
SP-1
y E
KLF
Es re
quer
ido
para
el c
ambi
o de
act
ivac
ión
de g
enes
de
la
glob
ina
en la
mad
urac
ión
ce
lula
r. A
ctúa
com
o un
act
ivad
or
cuan
do e
sta
unid
o al
pr
omot
or d
e ge
n de
la γ
-gl
obin
a
Act
úa c
omo
sile
ncia
dor
cuan
do s
e un
e al
sile
ncia
dor
de ε
-glo
bina
en
la p
rese
ncia
de
l fac
tor Y
Y1.
G
AT
A-2
D
escr
ito e
n hu
man
o (D
orfm
an, e
t al.
1992
)
GA
TA z
inc-
finge
r
Se e
xpre
sa e
n m
ayor
ca
ntid
ad e
n la
s cél
ulas
pr
ecur
sora
s de
hem
atop
oyes
is te
mpr
ana.
Esen
cial
par
a he
mat
opoy
esis
embr
ioni
ca.
Impo
rtant
e pa
ra la
di
fere
ncia
ción
de
eritr
ocito
s
FOG
C
arac
teriz
ado
en
Rat
ón (T
sang
, et a
l. 19
97)
Tien
e nu
eve
zinc
-fin
gers
Se u
ne a
GA
TA-1
N
o tie
ne s
itio
de u
nión
en
el D
NA
Se c
o-ex
pres
a co
n G
ATA
-1
en lo
s erit
roci
tos d
uran
te
el d
esar
rollo
em
brio
nario
Fu
nció
n en
hem
atop
oyes
is.
EK
FL
Car
acte
rizad
o en
hu
man
o. (W
ijger
de,
et a
l, 1
996)
Zinc
-fin
ger
Se u
ne c
on a
lta a
finid
ad
al e
lem
ento
CA
CC
C
loca
lizad
o a
-90
con
resp
ecto
al s
itio
de
inic
io d
e la
tra
scrip
ción
.
Se u
ne a
la c
aja
CA
CC
C co
n m
ayor
afin
idad
en
el
prom
otor
del
gen
β-
glob
ina
que
en e
l del
γ-
glob
ina,
sugi
riend
o qu
e es
ta in
volu
crad
o en
la
activ
ació
n de
l pro
mot
or
del g
en d
e la
glo
bina
ad
ulta
. EK
FL ll
eva
el c
ompl
ejo
de
repr
esió
n m
SIN
3a/H
DA
C
a la
regi
ón d
el g
en ε
-gl
obin
a.
Act
iva
el p
rom
otor
del
gen
de
la β
-glo
bina
. Es
timul
a la
form
ació
n de
5�
HS3
. Es
ta im
plic
ado
en e
l cam
bio
de g
lobi
na fe
tal a
glo
bina
ad
ulta
. A
usen
cia
de E
KFL
con
lleva
a
la p
erdi
da to
tal d
e lo
s siti
os
HS
en e
l pro
mot
or d
e β
-gl
obin
a y
a la
per
dida
del
sit
io 5
�HS3
de
LCR
.
CK
II
Ei
No
tiene
uni
ón a
l DN
A.
Fosf
orila
EK
FL
incr
emen
tand
o su
M
odifi
ca la
act
ivid
ad d
e va
rias p
rote
ínas
.

46

47
2.2.2.4.LCR y modificaciones de la cromatina
La cromatina ejerce control sobre la expresión genética adquiriendo la conformación
�abierta� o �cerrada�. La cromatina abierta, generalmente esta hiperacetilada y es
susceptible a DNasaI contrario a lo que sucede en la cromatina cerrada.
La acción que desempeña LCR en las modificaciones de la cromatina ha sido
estudiada a partir de mutaciones que ocurren naturalmente dentro del locus humano de
la β-globina y a partir de experimentos en los que utilizan ratones con transgenes de la
β-globina localizados ectópicamente, demostrando que LCR tiene un rol en la
remodelación de la cromatina.
Forrester y colaboradores (1990) ponen en evidencia el rol de LCR en modular la
estructura de la cromatina del locus de la β-globina en ciertas mutaciones propias de
la enfermedad humana talasemia. La talasemia hispana es causada por una deleción de
35kb incluyendo los 22kb de la región LCR. En los individuos con esta mutación la
cromatina del locus de la β-globina tiene conformación cerrada, resistente a digestión
con DNasa I y transcripcionalmente inactiva, demostrando, que la región LCR tiene
función para abrir la cromatina además de su rol directo en la activación del gen de la
globina.
Sin embargo, experimentos en ratón con el transgen y en líneas celulares (Epner, et al.
1998) sugieren que LCR no es necesario para establecer la conformación abierta de la
cromatina del locus y que funciona primordialmente como �enhancer� en la activación
de la transcripción de los genes de la globina. Lo anterior sugiere que LCR puede
participar en la activación transcripcional en un dominio abierto llevando factores de
trascripción adicionales a través de la interacción con el complejo transcripcional ya
formado.

48
Harju y colaboradores (2002) afirman que la contradicción en los estudios en
mutaciones en talasemias con los realizados en líneas celulares y ratones puede
deberse a diferencias en el tamaño de la deleción.
2.3. Evolución de los mecanismos de regulación de los genes de β-Globina
La forma como han divergido los clusters de la β-globina en los diferentes linajes tiene
una implicación directa en la forma como se regula la expresión del gen en cada uno
de estas.
Debido a que tanto en aves como en mamíferos los clusters de β-globina comparten
varias características de los elementos de regulación, se podría asumir que estos
controles del sistema de regulación se generaron antes de la divergencia entre
mamíferos y aves (Figura 4) por lo que las funciones de regulación de la expresión del
cluster tales como activar la trascripción, abrir el dominio y aislamiento se
encontrarían tanto en mamíferos placentarios como en aves. En los dos grupos, la
expresión de los genes es controlada tanto por elementos dístales (LCR) como
proximales. En las aves, por ejemplo, se describió un aislador en el extremo 5� (HS4)
que bloquea la actividad del �enhancer� (Chung, et al. 1997), un elemento similar ha
sido descrito en los mamíferos, el sitio hipersensitivo 5� HS5, el cual podría ser
homologo al aislador en aves, (Tanimoto, et al. 2003; Farrel, et al. 2002). Sin
embargo, estas características comunes a la regulación de los genes de β-globina en
aves y mamíferos no se ven reflejadas en las comparaciones de las secuencias del
DNA de los clusters (Hardison, 1998). Una investigación mas profunda en las
secuencias que rodean el cluster de la β-globina podría aclarar si el aislador es una
adaptación única para este locus en aves o si el sitio HS5 representa un elemento
homologo en mamíferos (Hardison, 2001).
Se han encontrado corriente arriba del gen ε-β- globina de los marsupiales secuencias
homologas al locus de la región de control LCR de β-globina presente en mamíferos

49
placentarios. Por otro lado, en el cluster del pollo se ha localizado un �enhancer�entre
los genes βA� y ε -globina. En ratones transgenicos esta secuencia �enhancer� imita la
acción del LCR a pesar de ser mas pequeña y de estar en un sitio diferente comparado
con el sitio de LCR en los mamíferos (Hardison, 2001). De existir un ancestro común
entre los �enhancers� en el cluster de β-globina en el pollo y los que se encuentran
dentro de la región LCR del cluster de los mamíferos, se esperaría que las secuencias
de DNA se hubieran conservado. Sin embargo, estudios realizados por Reitman y
colaboradores (1993) y por Hardison (1998) indican que los alineamientos de las
secuencias de regulación en los genes ρ-β-ε- globina de pollo y el gen β-globina de
humano no encuentran sitios apareables significativos a pesar de que estas secuencias
comparten sitios de unión a factores de transcripción. Por lo tanto, o bien los
elementos de regulación cis comparten un ancestro común pero han divergido tanto
que no es posible alinearlos o han aparecido independientemente en cada grupo
(evolución convergente) (Wheeler, et al. 2001; Hardison, 2001). Sin embargo, la
diferencia en las secuencias de regulación no representa que el mecanismo haya
tenido origen en ancestros diferentes; es decir que, los genes ortólogos no
necesariamente tendrán mecanismos de regulación conservados (Tanay, et al. 2005).
Debido a que las secuencias de regulación son cortas y degeneradas, pueden ocurrir
variaciones en estas manteniéndose los sitios de unión a factores de trascripción
comunes. Este hecho permite un escenario de evolución diferente para las secuencias
de regulación, escenario en donde es posible que nuevos patrones de expresión
coexistan temporalmente con patrones ancestrales. Esta redundancia temporal hace
posible las modificaciones graduales que ultimadamente generan nuevos mecanismos
de regulación (Tanay, et al. 2005). De otra forma, Mariño-Ramírez y colaboradores
(2006) postulan el modelo de evolución �circuitous evolution� para el mecanismo de
regulación de las histonas, el cual difiere de la evolución convergente y sugiere que al
divergir de un mecanismo de regulación ancestral, presente en el ultimo ancestro
común, el patrón de expresión de las proteínas se conserva mientras que los
mecanismos de regulación cambian drásticamente. Este modelo de evolución descrito

50
en histonas podría observarse también en la regulación de los genes de β-globina,
puesto en estos se ha conservado el patrón de expresión de acuerdo a la etapa del
desarrollo pero los elementos de regulación aparentemente han divergido, pues en
estos no hay secuencias homologas.
Aun hay mucho por conocer respecto a la conservación de secuencias de elementos de
regulación del gen de la β-globina. En los estudios de evolución de secuencias de
regulación, se ha notado que para muchos genes aunque se conserven las secuencias
de las proteínas que codifican no se conservan los mecanismos de regulación
transcripcional (Jensen, et al. 2006). Así mismo, se ha estimado que la tasa de
evolución de los elementos de regulación es diferente de la de las secuencias
codificantes, esto debido a la �flexiblilidad� de estas secuencias que permiten
sustituciones nucleotídicas sin alterar su función. Como resultado, el numero total de
cambios en las secuencias de regulación es mayor que en las secuencias codificadoras
y menor que en los intrones y otras secuencias intergénicas sin función reguladora
(Wray, et al. 2003). Jensen y colaboradores (2006) indican que los cambios en los
mecanismos de regulación se pueden dar en el orden de solo 100 millones de años
implicando que los modelos de regulación pueden diferir considerablemente entre
grupos que han divergido más tiempo.
Hardison (1998) indica que la divergencia del cluster de β-globina entre aves y
mamíferos sucedió hace aproximadamente 250 millones de años, por tanto, según el
postulado de Jensen y colaboradores (2006), no es sorprendente encontrar que en las
regiones de regulación para este cluster, en aves y mamíferos, no se observen
alineamientos significativos.

51
3. FORMULACION DEL PROBLEMA Y JUSTIFICACION
3.1. Formulación del problema
Los genes que codifican para las globinas están presentes en contextos genómicos
diferentes por lo que es de esperarse que presenten diferencias en los mecanismos de
regulación. Algunos estudios han comparado la organización de los genes del locus
de la β-globina en diferentes especies indicando que el gen podría ser parálogo en
algunos de los linajes, es decir que el análisis filogenético muestra que no siempre los
genes son descendientes de un gen ancestral común, como es el caso en las aves y los
mamíferos (Hardison, 2001).
Las secuencias codificantes de los genes de la β-globina así como en sus elementos de
regulación de la transcripción son el material para estudiar los patrones de evolución
de la familia génica y de los mecanismos de regulación de la misma, así, buscar dichas
secuencias es la materia prima para empezar a definir las diferencias en los
mecanismos de regulación de la expresión de esta familia génica.
3.2. Preguntas de la investigación
Este proyecto se aproxima a la respuesta de las siguientes preguntas:
• Cuántas secuencias de regulación de la expresión de los genes que codifican
para la β-globina se han conservado en diferentes especies de mamíferos y,
cuales factores de transcripción se unen a dichas secuencias?
• Se ha conservado el locus de la región de control (LCR) en estas especies?
• Que tantas de estas secuencias de regulación se han conservado entre las aves y
los mamíferos?

52
Se entiende por secuencias conservadas cuando estas se pueden alinear, es decir que el
orden de las bases coincide, asumiendo así que dichas secuencias desempeñan un rol
similar en las diferentes especies que las poseen.
3.3. Justificación de la investigación
El estudio de las secuencias de regulación del gen de la β-globina en diferentes
especies permitirá establecer patrones de evolución de los mecanismos de regulación
de la expresión de este gen.
Una de las características importantes en el mecanismo de regulación de este gen en
particular es que de este depende el cambio de la globina que se expresa según el
estado del desarrollo del organismo. Muchos estudios han tratado de interpretar los
factores que gobiernan la activación y represión de genes de un estado a otro del
desarrollo. El entendimiento de este proceso podría ser útil en la búsqueda de posibles
tratamientos a enfermedades como la anemia falciforme y la talasemia, que son dos
de las enfermedades hematopoyeticas mas comunes (Harju, et al, 2002).
Este proyecto es el primer paso hacia el estudio de los patrones evolutivos que han
dado lugar a los diferentes mecanismos de regulación de la expresión del gen de la β-
globina. Al colectar y ubicar las secuencias reportadas como sitios de unión a
factores de transcripción y las secuencias que han sido descritas como promotores,
�enhancers� o aisladores del locus de la β-globina se aporta la materia prima para
futuros estudios que permitan inferir los patrones que gobiernan la regulación de esta
familia de genes y establecer cómo dichos mecanismos han cambiado a través de la
evolución.

53
4. OBJETIVOS
4.1. Objetivo general
Identificar y localizar en el cluster las regiones de regulación de la expresión de la
familia de genes de β-globina en diferentes especies.
4.2. Objetivos específicos
I. Localizar la región genómica donde se ubica el locus de la β-globina en cada
especie incluida en el estudio y ubicar en los genomas de estas especies, las
secuencias de regulación que se encuentran conservadas.
II. Determinar cuales de las secuencias de regulación se encuentran conservadas
en otras especies.
III. Identificar cuales son los factores de transcripción que intervienen en la
regulación de la expresión de la familia de genes de β-globina.
5. MATERIALES Y MÉTODOS
En la figura 5 se resumen los métodos propuestos para alcanzar los objetivos.

54
Búsqueda de secuencias de regulación del grupo de genes de β-globina en varias especies
Búsqueda de secuencias reportadas en la Base de Datos TransFac®
Localización de secuencias de regulación en el genoma de diferentes especies
Generar Tabla de datos: Numero de accesión a la secuencia, coordenadas de la secuencia, factor de transcripción que se une, cromosoma, especie, enlace a artículo en Pubmed
Alineamiento del fragmento de DNA de ~1000bp en el genoma de la especie en la cual la secuencia de regulación había sido descrita
Ubicación de la secuencias de regulación.(Coordenadas en el cromosoma).
Búsqueda de secuencias conservadas
Humano, Chimpancé, Ratón, Rata, Perro y Pollo.
Generar archivo de texto en formato FASTA con las secuencias de regulación conservadas.
Ubicar cada gen de β globina en el genoma de varias especies
ENCODE Project: Anotaciones en el genoma (RefSequences)
Búsqueda de secuencias homologas a las reportadas en ENCODE
Blat con la secuencia de la proteína o predicciones de genes en HomoloGene cuando no hay Refseq.
Mapear el Locus Control Región (LCR) y otras regiones de regulación del grupo de genes de β globina reportadas en artículos.
Localización de los sitios hipersensitivos caracterizados por High-throughput (Dorschner et al.2004)
Localización de secuencias de regulación descritas experimentalmente.
Búsqueda de secuencias conservadas entre los sitios reportados en la literatura y descritos experimentalmente, usando la herramienta BLAT en el �UCSC Browser�
Ubicación de las secuencias de regulación conservadas de acuerdo a las coordenadas en el cromosoma.
Generar un mapa del cromosoma donde se ubica la familia de genes de β globina en cada especie ubicando también las secuencias de regulación y los sitios hipersensitivos (LCR).
Flanquear las secuencias de regulación reportadas en TransFac
Figura 5. Diagrama general de los métodos a seguir para alcanzar los objetivos
propuestos. Los recuadros rosados muestran el objetivo de la actividad y los recuadros
azules muestran los resultados esperados e indicadores. Ver protocolos 1 y 2 en
anexos.

55
5.1. Métodos para alcanzar el primer objetivo específico
5.1.1. Secuencias de regulación del locus de la β- globina
Se accedió a las secuencias de regulación del locus de la β- globina a través de la base
de datos TransFac® en la cual se encuentra información de factores de transcripción,
sus secuencias de unión y los genes que regulan. Los datos encontrados en TransFac®
tienen respaldo experimental (Matys, et al. 2006) por tanto, por cada dato allí
almacenado hay una o varias citas bibliográficas en las que se describe la
aproximación experimental a cada factor de trascripción y su sitio de unión. Al
compilar y organizar toda la información publicada acerca de la regulación de la
transcripción en eucariotas, esta base de datos pone a disposición gran cantidad de
información que es susceptible de análisis y predicciones computacionales. A Octubre
de 2006 se encontraban en TransFac® 6133 registros sobre factores de transcripción,
de los cuales 1040 han sido descritos experimentalmente en humano y 765 en ratón,
por nombrar solo algunos (Matys, et al. 2006). El acceso a la base de datos en línea es
libre a través de: http://www.gene-regulation.com/pub/databases.html Sin embargo,
para la realización de este proyecto se accedió a TransFac Professional® a través de
membresía del National Center for Biotechnologic Information (NCBI) en
http://www.biobase.de/cgi-bin/biobase/transfac/start.cgi.
Los datos en TransFac® están almacenados en tres tablas: FACTOR, SITE y GENE
(Matys, et al. 2006), la búsqueda se realizó en la tabla GENE, de modo que esta se
limitó a los factores de trascripción reportados para los genes que codifican para
globinas (Figura 6). De la interfase de resultados solo se recopilaron datos de aquellos
factores de transcripción que estuvieran relacionados con los genes que codifican para
proteínas β- globina.

56
Figura 6. Interfase de resultados en la base de datos TransFac® al someter a
búsqueda �Globin� en la tabla GENE de dicha base. http://www.biobase.de/cgi-
bin/biobase/transfac/start.cgi
Por cada secuencia de unión a un factor de transcripción se obtuvo en la base de datos
la siguiente información: Número de accesión en TransFac®; descripción, la cual hace
referencia al gen que esta siendo regulado; especie en la que el sitio de regulación fue
caracterizado; número de accesión a la secuencia de un fragmento de DNA
caracterizado para determinar experimentalmente el sitio donde se une el factor de
trascripción; coordenadas de la ubicación de la secuencia de regulación en dicho
fragmento; número de accesión a articulo en Pubmed; nombre del factor de
transcripción que se une a la secuencia; región en la que se ubica la secuencia (sitio
hipersensitivo, �enhancer� o promotor) y la secuencia del sitio de regulación (Figura
7).
A partir de la información obtenida en la base TransFac® fue posible localizar las
secuencias en el genoma de las especies así como realizar alineamientos para
determinar cuales de estas secuencias se encuentran conservadas en otras especies.

57
De otro modo, se realizó una revisión bibliográfica acerca del locus de la región de
control, �enhancers� y promotores del grupo de genes de β-globina. Algunos artículos
incluían número de accesión a secuencias y coordenadas de las mismas, datos
necesarios para poder ubicar dichas secuencias en el genoma de la especie en la que
había sido caracterizada.
Figura 7. Interfase de información del numero de accesión R01943 de TransFac®. El
número V00409 es la accesión a la secuencia caracterizada para localizar la región de
control. Las coordenadas (286:293) hacen referencia a la ubicación de la secuencia
GGCTGGGG la cual es el sitio de unión al factor de transcripción TFIID.
http://www.biobase.de/cgi-bin/biobase/transfac/start.cgi

58
5.2. Métodos para alcanzar el segundo objetivo específico
5.2.1. Obtención de secuencias para alinear
Con el fin de ubicar cada secuencia de regulación en el genoma de la especie en la
cual fue caracterizada, se hicieron alineamientos utilizando la herramienta BLAT
(Kent, 2002) disponible en �UCSC genome browser� en http://genome.ucsc.edu.
BLAT esta diseñado para alinear rápidamente secuencias de 40pb o más con un 95%
de similitud. En el alineamiento de secuencias de DNA con BLAT, la secuencia
sometida es contrastada con la secuencia de todo un genoma. Para que BLAT alinee
la secuencia con la del genoma es necesario que un mínimo de 11 pares de bases
consecutivas coincidan (Kent, 2002).
Las secuencias de regulación varían entre 5 a 20pb aproximadamente. Si fuera posible
alinear un fragmento de este tamaño con la secuencia de un genoma se obtendrían
múltiples sitos donde coincide la secuencia del fragmento con la del genoma. Por lo
anterior y para cumplir con los requerimientos de la herramienta BLAT (someter
secuencias de mínimo 40pb) fue necesario incluir en el alineamiento el DNA que
flanquea las secuencias de regulación. De modo que, cada secuencia de regulación fue
flanqueada con 500pb a lado y lado. Al usar fragmentos de 1010pb aproximadamente
se logra un alineamiento especifico reduciendo la posibilidad de obtener falsos
positivos.
Así, los fragmentos a alinear se obtuvieron a partir de las coordenadas de las
secuencias reportadas en TransFac®, usando el numero accesión de cada secuencia en
la interfase de �Entrez Nucleotide� que ofrece NCBI en http://www.ncbi.nlm.nih.gov.
�Entrez Nucleotide� es una base de datos de secuencias de DNA sometidas por
múltiples fuentes incluyendo GeneBank, RefSeq y PDB. La interfase �Sequence
viewer� de �Entrez Nucleotide� permite ver la secuencia en formato FASTA y
delimitar la extensión de la misma utilizando coordenadas. Por ejemplo, una de las

59
secuencias reportadas en TransFac® (número de accesión U01317) comprende mas de
60000pb, dentro de esta secuencia el sitio de unión a un factor de transcripción se
encuentra desde el nucleótido 4611 al 4615 (coordenadas reportadas en TransFac
4611:4615) lo cual representa una secuencia de regulación de 5 pares de bases. Para
obtener esta secuencia flanqueada, como se requiere, se resta al primer número de la
coordenada tanto como se quiera extender la secuencia corriente arriba y se suma al
segundo numero en la coordenada tanto como se quiera extender corriente abajo. Para
este caso especifico para flanquear la secuencia de regulación por 500 pares de bases a
lado y lado, en la ventana �Sequence viewer� se someten las coordenadas 4111 a 5115,
4111 que resulta de restar 500 al primer número de las coordenadas, y 5115 de sumar
corriente abajo 500pb en la segunda coordenada. Así la interfase de �Entrez
Nucleotide� mostrara en formato FASTA una secuencia de 1005pb del fragmento de
más de 60000pb. La secuencia de 1005pb será la que se someta a alineamiento contra
el genoma en el cual la secuencia fue caracterizada, para este ejemplo, contra el
genoma del humano (Ver Anexo 1: Protocolo para determinar las coordenadas de las
secuencias de regulación conservadas en varias especies).
5.2.2. Determinación de las coordenadas en cromosoma
Una vez obtenida la secuencia en formato FASTA del fragmento de ~1000 pb el
siguiente paso es alinearla en el genoma de la especie correspondiente, para esto la
secuencia se debe copiar en la interfase de la herramienta BLAT en UCSC �Genome
Browser� allí se debe seleccionar el genoma en el que se desea buscar la secuencia
además de indicar el ensamble a usar según la especie. En la tabla 3 se indica el
ensamble usado en cada especie incluida en este estudio.

60
Tabla 3. Lista de ensambles de genomas usados para ubicar las secuencias de
regulación en el respectivo cromosoma de cada especie.
Especie Cromosoma Ensamble del Genoma
Humano 11 hg18 (Mar2006) Chimpancé 11 panTro2 (Mar2006) Ratón 7 mm8 (Feb2006) Rata 1 rn4 (Nov2004) Perro 21 canFam2 (May2005) Pollo 1 galGal3 (May2006)
Al someter las secuencias al alineamiento se obtiene una lista de resultados ordenados
de acuerdo al porcentaje de similitud. Esta interfase de resultados muestra, además del
porcentaje de similitud, el tamaño del fragmento que pudo ser alineado, es importante
tener en cuenta desde que nucleótido fue encontrada la secuencia y de esto depende el
cálculo de las coordenadas de la secuencia de regulación. En el caso ideal, el
fragmento sometido es alienado en su totalidad entonces, para calcular las
coordenadas del sitio de regulación, a la primera coordenada se suman 500, o la
cantidad de pares de bases que hayan sido agregadas al flanquear la secuencia,
haciendo esto, se esta retirando el fragmento que flaqueaba la secuencia corriente
arriba, de forma similar, a la segunda coordenada se debe restar 500 o el numero de
pares de bases que corresponden a la región que flanquea la secuencia de interés. De
esta forma se determinaron las coordenadas de cada sitio de regulación (Figura 8). El
mismo procedimiento se realizó con las secuencias reportadas en artículos donde
incluían el número de accesión a la secuencia y las coordenadas.
5.3. Métodos para alcanzar el tercer objetivo especifico
5.3.1. Ubicación de los genes de β-globina en cada especie
Cada sitio de regulación colectado fue localizado en un diagrama del cromosoma de
cada especie. Para entender mejor la posición de estos sitios respecto a la de cada gen
de la familia de genes de β globina, fue necesario primero ubicar las coordenadas de

61
Cromosoma
Fragmento donde se encuentra la secuencia de regulación según numero de accesión reportado en TransFac
2 Flanquear la secuencia de interés extendiendo las coordenadas 500 pb a
3 Obtener la secuencia del fragmento de interés más la
5
4Alinear el fragmento con la secuencia de interés en el genoma de la especie donde fue caracterizada.
5
5Al alinear se determinan las coordenadas a las que corresponde el fragmento en el cromosoma.
6
4
3
122
-500 +500
+500 -500
Localización de la secuencia de interés según coordenadas locales a un determinado fragmento de DNA
1
6Calcular las coordenadas de la secuencia de interés a partir de las coordenadas del fragmento alineado
estos genes en cada especie bajo estudio. Para este fin se consultaron las anotaciones
en �UCSC ENCODE Project� para humano, chimpancé, ratón y pollo, cuyos genomas
tienen varios genes anotados (Figura 9). Posteriormente, para localizar los genes en el
genoma de rata y perro se realizaron alineamientos BLAT con la secuencia de la
proteína codificada por cada gen en humano, buscando con esto el sitio homologo en
el otros genomas. Además de los alineamientos BLAT usando las secuencias de las
proteínas, se consulto el enlace HomoloGene para cada gen β globina anotado en
humano. HomoloGene es un sistema automático para la detección de homólogos entre
los genes anotados en varios genomas eucariotas completamente secuenciados, esta
herramienta esta disponible en la interfase de �Entrez Nucleotide� y a partir de la
predicción computacional muestra donde se ubicaría en otras especies el gen
homologo al que esta siendo consultado. HomoloGene esta disponible en línea en:
http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=homologene.
Figura 8. Diagrama general de los métodos para determinar las coordenadas de las
secuencias de regulación.

62
De esta forma, con base en las anotaciones genómicas en �UCSC ENCODE Project�,
los alineamientos recíprocos de las secuencias de las proteínas y las predicciones de
HomoloGene, se pudo determinar la ubicación de cada gen del grupo de genes de β-
globina en cada especie bajo estudio (Ver Anexo 2: Protocolo para la ubicar los genes
de la familia de β-globina en el genoma de cada especie incluida en el estudio).
Figura 9. Interfase �UCSC ENCODE Project� de la región ENm009 que corresponde
a la familia de genes de la β-globina. Se muestran los genes HBE1, HBG2, HBG1,
HBD y HBB, que corresponden a ε-Gγ-Aγ-δ-β-globina respectivamente.
5.4. Métodos para alcanzar el cuarto objetivo específico
5.4.1. Secuencias de regulación conservadas
Usando la herramienta �Genome Browser� de USCS http://genome.ucsc.edu es posible
conocer si la secuencia de regulación se encuentra conservada en otras especies. Para
este fin se activa la opción �conservation�, seleccionando �Full� en esta entrada bajo
�Comparative Genomics� en la interfase �Genome Browser� (Figura10) habiendo
activado esta opción, al someter las coordenadas de la secuencia de regulación este
�Browser� realizara un alineamiento múltiple de la secuencia en el genoma de varias
especies y mostrara en cuales ha logrado alinearla asumiendo con esto que la
secuencia se ha conservado en ellas presentando una función similar.

63
Una vez sometidas las coordenadas de la secuencia de regulación, si esta se ha
conservado en otras especies se obtiene una lista de las especies donde se pudo alinear
la secuencia y frente a cada especie la secuencia encontrada (Ver Anexo 1 paso 7),
para ampliar la información y acceder a las coordenadas de la secuencia en cada
especie se hace clic en la lista de secuencias conservadas con lo cual se abre una nueva
interfase que ofrece un enlace para cada secuencia en �Genome Browser� de donde se
obtienen las coordenadas para esa secuencia en cada especie donde se encontró
conservada (Ver Anexo 1 paso 8 y 9).
Figura 10. Sección de la interfase �Genome Browser� donde se activa la opción para
acceder a la información sobre conservación. http://genome.ucsc.edu
5.4.2. Secuencias en formato FASTA
A partir de los datos obtenidos en la base de datos TransFac® se accedió a varias
secuencias de regulación y se ubicaron en el genoma de la especie en donde dichos
sitios habían sido caracterizados, luego se determinó cuales de las secuencias se han
conservado en otras especies. El siguiente paso fue colectar las secuencias en formato
FASTA de modo que puedan luego ser agrupadas según el factor de transcripción que
se une a estas y establecer comparaciones.

64
Con el fin de obtener las secuencias en formato FASTA, estando en la interfase de el
alineamiento múltiple en especies que tienen la secuencia conservada, se hace clic en
D que hace referencia a DNA, al hacer esto se abre una nueva interfase que muestra
las coordenadas donde se localiza la secuencia en la especie y la secuencia de
nucleótidos en el formato deseado (Figura 11). Cada secuencia fue copiada en un
archivo de texto.
Figura 11 A. Interfase del alineamiento múltiple bajo la opción de conservación en
�USCS Genome Browser� Al hacer clic en la D para cada especie en la lista, se
obtienen las coordenadas homologas a las del humano en las otras especies.
B. Interfase para acceder al DNA a partir del alineamiento múltiple. Los corchetes y el
texto azul no hacen parte de la interfase http://genome.ucsc.edu.
A
Coordenadas en el genoma del ratón que corresponden al sitio de regulación homologo al de las coordenadas en el humano.
B

65
5.5. Métodos para alcanzar el quinto objetivo específico
5.5.1. Agrupación de secuencias según factor de transcripción
Una vez obtenidas todas las secuencias de regulación en las diferentes especies estas
secuencias se clasificaron según el factor de transcripción que a estas se une.
Posteriormente, se crea un archivo de texto por cada factor de transcripción, cada
archivo contiene las secuencias de unión para un factor de transcripción determinado.
Se genera también una tabla donde se recopilan las coordenadas organizadas según el
factor de transcripción que se une y finalmente se genero una lista de factores de
transcripción que intervienen en la regulación de la familia de genes de β-globina.
6. RESULTADOS Y DISCUSIÓN
6.1. Colección de sitios de regulación
Se colectaron 84 sitios de regulación a partir de la base de datos TransFac®, de esas
84 secuencias, 31 fueron descritas experimentalmente en humano, 24 en ratón, 25 en
pollo y 4 en pato, estas ultimas 4 secuencias no fueron incluidas en el estudio debido a
que no hay secuencia completa del genoma del pato por tanto no era posible
determinar coordenadas en este genoma para estas secuencias. Los números de acceso
y la información pertinente a estos sitios de regulación se encuentran en el anexo 3
(Sitios de regulación obtenidas a partir de la base de datos TransFac®). Las secuencias
de los sitios colectados se registran en el anexo 8 (Secuencias en formato FASTA de
los sitios de regulación colectados).
De otra forma, a partir de la revisión bibliográfica se encontró información sobre
múltiples sitios de regulación caracterizados en humano, ratón y pollo, pero de la
descripción de estos sitios solo fue posible localizar aquellos en donde el artículo
incluía además del número de accesión a la secuencia, las coordenadas para su

66
ubicación. Así, a partir de la revisión bibliográfica, se mapearon 37 secuencias
caracterizadas en humano y 5 en pollo, y de estas se determinó cuales se han
conservado en las otras especies bajo estudio (humano, chimpancé, ratón, rata, perro,
pollo) la información de estos sitios se encuentra en el anexo 4 (Sitios de regulación
obtenidos a partir de la revisión bibliográfica)
6.1.1. Factores de transcripción
Del total de sitios de regulación colectados hay secuencias de unión a 24 factores de
transcripción. En el anexo 7 se organizan las coordenadas de los sitios colectados de
acuerdo al factor de transcripción que se une a estos. En la tabla 4 se indica el número
de secuencias colectadas para cada factor de transcripción.
Según la revisión de Harju y colaboradores (2002) los factores de transcripción, FOG,
EKLF, CKII, FKLF, DRED, COUP-TFII (Tabla 2) son propios de la regulación de la
expresión del cluster de genes de β-globina pero en las secuencias colectadas en este
estudio, no hay sitios para la unión a estos factores. Esto indica que a pesar de que han
sido identificados los factores de transcripción que intervienen en la regulación del
cluster, aun hace falta información para la localización de sus secuencias de unión en
los genomas en donde han sido caracterizados.
Tabla 4. Número de secuencias colectadas por factor de transcripción
Factor de transcripción No. de secuencias Factor de transcripción No. de secuencias AP1 6 NF-E2 16 B1 14 NFI-CTF 3 B2 13 Nrf 5
BP1 19 Nrf1 6 BP2 17 Nrf2 6
CAC-Binding protein 15 POU2F1 7 CACC-Binding factor 12 PPUR 5
CACD 6 PPYR 5 C-Fos 5 Sp1 12 C-Jun 5 TBP 5 Cp2a 5 TFIID 14
GATA-1 140 MafG 5

67
6.2. Patrón de conservación de los sitios de regulación colectados
Se colectaron un total de 122 sitios de regulación de los cuales 68 habían sido
caracterizados en humano, 24 en ratón y 30 en pollo. Se determinó si cada uno de
estos sitios estaba conservado en otras especies.
Los resultados de los alineamientos para cada sitio de regulación mostraron posibles
secuencias conservadas en diferentes especies, solo se incluyeron en el estudio
aquellas especies cuyos genomas han sido secuenciados completamente de modo que
se contara con la información suficiente para poder determinar si las coordenadas
donde cada secuencia de regulación estaba siendo alineada correspondía al sitio
genómico donde se ubica el cluster de la β-globina. Así por ejemplo, se encontró que
algunas secuencias estaban conservadas en zarigüeya y Xenopus, pero las coordenadas
donde se alineaban los sitios de regulación en estos genomas hacían referencia a
�scaffold� y no a un cromosoma definido. Por lo anterior, se incluyen en este estudio
las siguientes especies y no otras: humano, chimpancé, ratón, rata, perro y pollo.
El patrón de conservación de los sitios de regulación en las seis especies incluidas en
el estudio se recopila en la tabla 5.
La cantidad de datos obtenidos en mamíferos y aves varia drásticamente, de los sitios
de regulación obtenidos a través de TransFac® un promedio de 59 sitios fueron
colectados por cada mamífero mientras que en aves solo se obtuvieron 35 sitios en
total. Es de notar, sin embargo, que el locus de la β-globina ha sido ampliamente
caracterizado en ambos grupos, pero en las aves, solo se cuenta con secuencias
caracterizadas en pollo mientras que en mamíferos las hay en humano y en ratón.
Así, la diferencia en el número total de secuencias por especie además de mostrar que
existe una mayor disponibilidad de información del locus de β-globina en mamíferos,
pone en evidencia un patrón de conservación donde la mayoría de secuencias
caracterizadas en aves no están conservadas en mamíferos y viceversa.

68
De las secuencias caracterizadas en humano y ratón la mayoría están conservadas en
los otros mamíferos incluidos en el estudio (Figura 12).
Tabla 5. Patrón de conservación de las secuencias colectadas.
Numero de secuencias conservadas en: Secuencias a partir de TransFac® Humano Chimpancé Ratón Rata Perro Pollo
Humano (31)
31 31 28 29 29 4
Ratón (24)
22 22 24 22 21 1
Pollo (30)
10 10 9 8 0 30
Sub. Total 63 63 61 59 50 35 Numero de secuencias conservadas en: Secuencias a parir de
Revisión literatura Humano Chimpancé Ratón Rata Perro Pollo Humano
(37)
37
37
30
28
26 1
Pollo (5)
0
0
0
0
0
5
Total de secuencias localizadad en cada
genoma
100
100
91
87
76
41
Numero de secuencias conservadas entre las especies bajo estudio
05
101520253035
Humano Ratón Pollo
Especie en la que se caracterizaron las secuencias de regulacion
Num
ero
de s
itios
pre
sent
es Sitios conservados en
Humano
Sitios conservados enChimpance
Sitios conservados enRaton
Sitios conservados enRata
Sitios conservados enPerro
Sitios conservados enPollo
Figura 12. Número de secuencias conservadas por especie. Es de notar que de las
secuencias caracterizadas en mamíferos muy pocas están presentes en el ave, así
mismo, de las caracterizadas en pollo pocas están conservadas en los mamíferos. En
esta grafica solo se incluyen las secuencias obtenidas a partir de TransFac.

69
Existen varias secuencias disponibles del cluster de β-globina tanto en aves (pollo)
como en mamíferos (humano) y se conocen varias similitudes entre los clusters de
estos dos grupos. En ambos, aves y mamíferos, los genes de β-globina se expresan
únicamente en los eritrocitos y de forma especifica a la etapa del desarrollo. Solo en
los eritrocitos este cluster de genes se encuentra en un dominio de cromatina abierto.
Además, tanto en aves como en mamíferos, la expresión de los genes de β-globina es
controlada tanto por secuencias de regulación proximales como dístales. A pesar de
estas semejanzas, entre aves y mamíferos, en organización y regulación de este grupo
de genes, al comparar las secuencias completas de los clusters de β-globina en las dos
especies Reitman y colaboradores (1993) encontraron que las similitudes están
restringidas a las regiones codificantes en los genes. De forma similar, Hardison
(1998) realizó alineamientos de las secuencias de los genes de β-globina en humano y
pollo concluyendo que cada gen de β-globina en humano esta igualmente distanciado
de cualquier gen de β-globina en pollo, es decir, el gen ε-globina en humano no esta
mas cercanamente relacionado con el gen embrionario ε-globina de pollo que con el
gen de la edad adulta β-globina en pollo.
Estas diferencias observadas a nivel de las secuencias de los genes (Hardison, 1998) y
de las zonas intergénicas (Reitman, et al. 1993) concuerdan con lo observado con las
secuencias de regulación colectadas. La mayoría de las secuencias caracterizadas en
mamíferos no están presentes en pollo. Contrario a lo que se pensaba antes de la
secuenciación de los genes, Hardison (1998, 2001) y otros autores coinciden al afirmar
que los clusters de β-globina en aves y mamíferos son parálogos es decir que no
comparten el mismo gen ancestral.
6.3. Localización de los genes del cluster de β-globina en las diferentes especies
incluidas en el estudio
En humano hay cinco genes descritos en el cluster de genes de β-globina, en su orden
ε-γG-γA-δ-β corresponden a las anotaciones HBE1, HBG2, HBG1, HBD, HBB en

70
�UCSC ENCODE Project� de estos cinco genes se localizaron cinco homólogos en
chimpancé HBE1, PanHBG1, HBG1, HBD, HBB, cuatro en ratón: hbb-y, hbb-bh1,
hbb-b1, hbb-b2 que corresponden en ese orden a los genes ε-γ-δ-β en humano. Por su
parte, en el genoma de la rata, con base en anotaciones genómicas y predicciones de
HomoloGene, se mapearon cinco genes: Hbe1, Hbe2 (predicción), Hbg1, MGC72973
beta-glo y Hbb los cuales son homólogos a los genes ε-γ-γ-δ-β en humano. En el caso
del perro, debido a que su genoma no esta tan bien anotado como los de las otras
especies incluidas en este estudio, no se encontraron secuencias de referencia para el
locus de β globina por lo que se ubicaron 5 genes con base en predicciones de la
herramienta HomoloGene, estos son: LOC485256, LOC485255, LOC609402,
LOC480704 y LOC476825 los cuales son homólogos a los genes ε-γ-β-β-β en
humano. Las coordenadas para cada uno de los genes en las diferentes especies se
encuentran en la tabla 6.
De acuerdo a las coordenadas obtenidas para cada gen, en los mamíferos todos los
genes se localizan cerca a las secuencias de regulación colectadas. Por el contrario, en
el cromosoma 1 del pollo se mapearon 6 genes, de los cuales tres son predicciones de
HomoloGene y se ubican cerca a las únicas cuatro secuencias de regulación que
habiendo sido descritas en mamíferos son homologas en pollo (Tabla 5). Los otros tres
genes son anotaciones en UCSC ENCODE Project, y estos se ubican en otra región
en el cromosoma 1, la cual coincide con el sitio donde se ubican todas las secuencias
de regulación caracterizadas en pollo.
La estructura típica de los genes de la familia de la β-globina es 2 intrones y tres
exones (Hardison y Miller, 1993), todos los genes mapeados en este estudio presentan
dicha estructura exceptuando las predicciones de HomoloGene en Perro y en pollo.
Otra diferencia para señalar es que en todos los mamíferos el cluster de β-globina se
encuentra en la cadena negativa mientras que en el pollo esta en la cadena positiva.

71
Tab
la 6
. Coo
rden
adas
de
los g
enes
del
clu
ster
de β
glob
ina
en la
s es
peci
es in
clui
das
en e
l est
udio
. Se
regi
stra
el n
ombr
e ba
jo e
l cua
l se
encu
entra
la
anot
ació
n en
UC
SC E
NC
OD
E Pr
ojec
t, el
ens
ambl
aje
del
geno
ma
al c
ual
corr
espo
nden
las
coo
rden
adas
o s
i es
una
pr
edic
ción
de
Hom
oloG
ene,
así
com
o la
est
ruct
ura
del g
ene
(intro
nes y
exo
nes)
y su
sent
ido
en e
l cro
mos
oma
(cad
ena
-/+)
Es
peci
es
G
en
Hum
ano
Cr.
11
Chi
mp.
Chr
. 11
Rat
on C
hr.7
R
ata
Chr
. 1
Perr
o C
hr. 2
1 Po
llo C
hr.1
H
BE
1 (R
efS
eq M
ar20
06)
5246
156-
5247
949
2 In
trone
s / 3
Exo
nes
Min
us s
trand
HB
E1
(Ref
Seq
Mar
2006
) 50
7541
4-50
7720
9 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
Hbb
-y
(Ref
Seq
Feb
2006
) 10
3725
577-
1037
2702
8 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
Hbe
1 pr
edic
ted
(R
efS
eq N
ov20
04)
1616
5142
3-16
1652
702
2 In
trone
s / 3
Exo
nes
Min
us s
trand
LOC
4852
56
(Hom
oloG
ene)
31
3213
90-3
1331
274
4 In
trone
s / 5
Exo
nes
Min
us s
trand
HB
E1
(R
efS
eq M
ay20
06)
1994
3357
3-19
9434
763
2 In
trone
s / 3
Exo
nes
Plus
stra
nd
ε
PanH
BG
1 (R
efS
eq M
ar20
06)
5075
534-
5076
958
2 In
trone
s / 3
Exo
nes
Min
us s
trand
H
BE
1 (H
omol
oGen
e)
1881
2473
1-18
8127
250
3 In
trone
s / 3
Exo
nes
Plus
stra
nd
γG
HB
G2
(R
efS
eq M
ar20
06)
5230
997-
5232
587
2 In
trone
s / 3
Exo
nes
Min
us s
trand
Hbe
2 Pr
edic
ted
(Ref
Seq
Nov
2004
) 16
1647
652-
1616
4927
9 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
H
BG
2 (H
omol
oGen
e)
1881
3414
6-18
8137
740
4 In
trone
s / 5
Exo
nes
Plus
stra
nd
γ
A
HB
G1
(Ref
Seq
Mar
2006
) 5,
226,
078-
5,22
7,66
3 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
HB
G1
(Ref
Seq
Mar
2006
) 50
6025
5-50
6184
4 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
Hbb
-bh1
(R
efS
eq F
eb20
06)
1037
1545
9-10
3716
983
2 In
trone
s / 3
Exo
nes
Min
us s
trand
Hbg
1 (R
efS
eq N
ov20
04)
1616
4035
9-16
1641
737
2 In
trone
s / 3
Exo
nes
Min
us s
trand
LOC
4852
55
(Hom
oloG
ene)
31
3112
48-3
1316
056
3 In
trone
s / 4
Exo
nes
Min
us s
trand
LOC
4281
14 b
eta-
h (R
efS
eq M
ay20
06)
1994
3693
8-19
9438
081
2 In
trone
s / 3
Exo
nes
Plus
stra
nd
δ
HB
D
(Ref
Seq
Mar
2006
) 52
1063
5-52
1243
4 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
HB
D
(Ref
Seq
Mar
2006
) 50
3909
9-50
4089
6 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
Hbb
-b1
(Ref
Seq
Feb
2006
) 10
3686
347-
1036
8774
4 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
MG
C72
973
beta
-glo
(R
efS
eq N
ov20
04)
1615
9067
1-16
1598
127
2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
HB
B
(Ref
Seq
Mar
2006
) 52
0327
2-52
0487
7 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
HB
B
(Ref
Seq
Mar
2006
) 50
2937
4-50
3097
9 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
Hbb
-b2
(Ref
Seq
Feb
2006
) 10
3686
345-
1036
8772
4 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
Hbb
(R
efS
eq N
ov20
04)
1615
8485
8-16
1620
193
2 In
trone
s / 3
Exo
nes
Min
us s
trand
LOC
4807
84
(Hom
oloG
ene)
31
2993
85-3
1300
913
2 In
trone
s / 3
Exo
nes
Min
us s
trand
HB
B
(Ref
Seq
May
2006
) 19
9436
906-
1994
4199
9 2
Intro
nes
/ 3 E
xone
s Pl
us s
trand
LO
C60
9402
(H
omol
oGen
e)
3130
5498
-313
0724
4 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd
HB
B
(Hom
oloG
ene)
18
8131
689-
1881
3339
2
2 In
trone
s / 3
Exo
nes
Plus
stra
nd
Β
LOC
4768
25 *
(H
omol
oGen
e)
3129
7094
-312
9835
5 2
Intro
nes
/ 3 E
xone
s M
inus
stra
nd

72
En el humano la organización de los genes en el cromosoma coincide con el orden en
que se expresan durante el desarrollo (figura 2). Así, el primer gen en expresarse, el
gen ε-globina, se encuentra mas cerca al extremo 5� del cluster y el ultimo en
expresarse, β-globina mas cerca del extremo 3�. Hardison y Miller (1993) describen
que en todos los mamíferos el gen ε-globina se ubica hacia el extremo 5� del cluster y
solo se expresa en los glóbulos rojos embrionarios.
Hardison y Miller (1993) han estimado que en la evolución del cluster de genes de β-
globina, hubo una duplicación del gen γ-globina en los primates antropoides,
duplicación que coincide con la aparición de un nuevo patrón de expresión en este gen
γ el cual se expresa en la etapa fetal en los mamíferos antropoides mientras que en la
mayoría de especies la expresión del gen γ-globina esta limitada a la etapa
embrionaria. El surgimiento de este nuevo patrón de expresión en los genes γ-globina
coincide aproximadamente con la duplicación del gen la evolución de los primates.
Según lo anterior se esperaba encontrar dos genes gamma en humano y chimpancé, sin
embargo, solo un gen gamma fue localizado en chimpancé. Únicamente en rata se
encontró un gen homologo al HBG2 (γG�globina) de humano, mientras que en los
otros mamíferos y en pollo, solo se ubico un gen gamma que corresponde al homologo
al HBG1 (γA�globina) en humano.
Respecto al cluster de genes en rata, Okumura y colaboradores (2004) describen que
hay un alto grado de variación en este cluster en rata al compararlo con el de otros
mamíferos. El sistema de hemoglobina en la rata es extremadamente heterogéneo
(Satoh, et al. 1999). Se han descrito cuatro proteínas de β- globina en la rata adulta y
dos en la etapa fetal (ε1, ε3-globina). Así, la expresión de dos proteínas de globina en
el feto de la rata justifica la presencia de dos genes gamma en este genoma.
Alineamientos de las secuencias de DNA de los genes en rata muestran una alta
similitud entre los genes gamma (γ1-γ2) (8.6% de divergencia) (Satoh, et al. 1999), por

73
lo anterior se puede sugerir que este gen surgió en rata a partir de una duplicación
como es el caso del humano.
Surge entonces la inquietud de por que en rata hay dos genes fetales, mientras que en
ratón solo hay uno, siendo la rata y el ratón dos especies cercanas evolutivamente. La
ausencia de duplicación del gen gamma en ratón se puede explicar con la expresión
temprana del gen β-globina en ratón. Hardison y Miller (1993) han descrito que este
gen se expresa en la etapa fetal en ratón, conejo y galago mientras que en el resto de
mamíferos su expresión se inicia en la etapa adulta. La expresión del gen beta durante
la etapa fetal en el ratón, justificaría la ausencia en este genoma de un segundo gen
gamma cuya expresión es fetal.
La expresión fetal del gen beta permite a Hardison y Miller (1993) inferir que el
estado ancestral del gen β-globina es iniciar su expresión en la etapa fetal y que el
aparecimiento del gen γ-globina y su expresión fetal en primates antropoides retardo la
expresión del gen β-globina hasta la etapa adulta en estas especies.
En el cluster de β-globina en pollo, se han descrito 4 genes: ρ-β-β-ε (Reitman, et al.
1993), en este estudio se localizaron seis genes, tres de los cuales son predicciones de
HomoloGene y se ubican en una región diferente a la de los genes que están anotados
en ENCODE. Reitman y colaboradores (1993) realizaron comparaciones de la
secuencia de los genes de β-globina en aves para deducir el esquema de su evolución.
Ellos determinaron que en aves el cluster de β-globina se formó a partir de tres
duplicaciones genéticas y por lo menos 4 conversiones. Los tres genes ubicados
corriente arriba de los genes anotados por ENCODE Project, tienen el orden reportado
en la literatura (ρ-β-β-ε) pero solo se ubico un gen β. De los genes mapeados, los tres
obtenidos a través de HomoloGene podrían ser resultado de una duplicación debido a
que cerca de estos genes se ubican secuencias de regulación conservadas y a que su
orden coincide con el descrito por Reitman y colaboradores (1993) sin embargo,
también podría tratarse de pseudo genes, debido a que son predicciones

74
computacionales y a que están muy alejados del sitio donde se ubicaron las secuencias
de referencia de ENCODE. Además se localizaron 6 genes en pollo y, para esta
especie, solo se han reportado 4 genes.
6.4. Ubicación de los sitios de regulación en el genoma de cada especie
De acuerdo a la tabla 4, 100 sitios de regulación fueron ubicados en el genoma de
humano y chimpancé, 91 en ratón, 87 en rata, 76 en perro y 41 en pollo. El anexo 5,
muestra las coordenadas en cada especie para cada sitio de regulación. Las figuras 13,
14, 15, 16, 17 y 18 muestran el mapeo de estos sitios y de los genes del cluster de β-
globina en Humano, chimpancé, ratón, rata, perro y pollo respectivamente.
En la revisión bibliográfica varias de las secuencias obtenidas hacen referencia a los
sitios hipersensitivos a DNasa (HS) así como a los promotores y �enhancers�
proximales a los genes. Hay entonces, información redundante respecto a cada HS, a
los promotores y �enhancers� proximales (Tabla 7), y no hay coincidencia entre los
sitios caracterizados por Dorschner y colaboradores (2004) a través de �High
throughout put� y los caracterizados por los otros autores. Se considera que el margen
de error en el trabajo de Dorschner, et al. (2004) es mayor respecto a los otros estudios
citados; por tratarse de una caracterización sistematizada que abarca una gran
extensión de DNA (~300 Kb) mientras que, en las descripciones de los otros autores la
aproximación es experimental y solo hacia uno o dos sitios en particular. Por esta
razón, el mapa definitivo de los sitios de regulación seria sin incluir las regiones
caracterizadas a través de �High-throughput� de esta forma también se reduce la
redundancia de información.

75
Figu
ra 1
3. D
iagr
ama
del c
lust
er d
e β-
glob
ina
en h
uman
o. L
os s
itios
des
crito
s en
hum
ano
se m
uest
ran
en ro
jo, e
n az
ul lo
s de
scrit
os e
nra
tón
y co
nser
vado
s en
hum
ano
y en
ver
de l
os s
itios
car
acte
rizad
os e
n po
llo y
pre
sent
es e
n hu
man
o. L
as r
egio
nes
de l
a re
visió
nbi
blio
gráf
ica
se m
uest
ran
sobr
e la
lín
ea d
el c
rom
osom
a en
neg
ro y
en
colo
res
que
son
igua
les
en t
odas
las
esp
ecie
s pa
ra d
enot
arho
mol
ogía
.

76
Figu
ra 1
4. D
iagr
ama
del c
lust
er d
e β-
glob
ina
en C
him
panc
é. L
os s
itios
des
crito
s en
hum
ano
se m
uest
ran
en ro
jo, e
n az
ul lo
s des
crito
s en
rató
n y
en v
erde
los
sitio
s ca
ract
eriz
ados
en
pollo
y p
rese
ntes
en
chim
panc
é. L
as re
gion
es d
e la
rev
isió
n bi
blio
gráf
ica
se m
uest
ran
sobr
e la
líne
a de
l cro
mos
oma
en n
egro
y e
n co
lore
s que
son
igua
les e
n to
das l
as e
spec
ies p
ara
deno
tar h
omol
ogía
.

77
Figu
ra 1
5. D
iagr
ama
del c
lust
er d
e β-
glob
ina
en R
atón
. Los
siti
os d
escr
itos
en ra
tón
se m
uest
ran
en a
zul,
los
desc
ritos
en
hum
ano
en
rojo
y e
n ve
rde
los
sitio
s ca
ract
eriz
ados
en
pollo
y p
rese
ntes
en
rató
n. L
as r
egio
nes
de la
rev
isió
n bi
blio
gráf
ica
se m
uest
ran
sobr
e la
lín
ea d
el c
rom
osom
a en
neg
ro y
en
colo
res q
ue so
n ig
uale
s en
toda
s las
esp
ecie
s par
a de
nota
r hom
olog
ía.

78
Figu
ra 1
6. D
iagr
ama
del c
lust
er d
e β-
glob
ina
en R
ata.
Los
siti
os d
escr
itos
en h
uman
o se
mue
stra
n en
roj
o, e
n az
ul lo
s de
scrit
os e
n ra
tón
y en
ver
de lo
s sit
ios
cara
cter
izad
os e
n po
llo y
pre
sent
es e
n ra
ta. L
as r
egio
nes
de la
rev
isió
n bi
blio
gráf
ica
se m
uest
ran
sobr
e la
lín
ea d
el c
rom
osom
a en
neg
ro y
en
colo
res q
ue so
n ig
uale
s en
toda
s las
esp
ecie
s par
a de
nota
r hom
olog
ía.

79
Figu
ra 1
7. D
iagr
ama
del c
lust
er d
e β-
glob
ina
en P
erro
. Los
siti
os d
escr
itos
en h
uman
o se
mue
stra
n en
roj
o, e
n az
ul lo
s de
scrit
os e
n ra
tón
y en
ver
de lo
s sit
ios
cara
cter
izad
os e
n po
llo y
pre
sent
es e
n pe
rro.
Las
regi
ones
de
la re
visió
n bi
blio
gráf
ica
se m
uest
ran
sobr
e la
lín
ea d
el c
rom
osom
a en
neg
ro y
en
colo
res q
ue so
n ig
uale
s en
toda
s las
esp
ecie
s par
a de
nota
r hom
olog
ía.

80
Figu
ra 1
8. D
iagr
ama
del c
lust
er d
e β-
glob
ina
en P
ollo
. Los
siti
os d
escr
itos
en p
ollo
se
mue
stra
n en
ver
de, l
os d
escr
itos
en h
uman
o en
ro
jo y
en
verd
e lo
s siti
os c
arac
teriz
ados
en
hum
ano
y pr
esen
tes e
n po
llo. L
as re
gion
es d
e la
revi
sión
bib
liogr
áfic
a se
mue
stra
n so
bre
la
línea
del
cro
mos
oma
en n
egro
.

81
Tabla 7. Número de secuencias colectadas y localizadas por región de regulación, a
través de la revisión bibliográfica.
Número de secuencias colectadas y ubicadas en el cluster
Región Humano Chimpancé Ratón Rata Perro Pollo
HS5 2 2 2 1 1 0
HS4 2 2 2 2 2 1
HS3 4 4 4 4 4 0
HS2 2 2 2 2 2 0
HS1 3 3 3 3 3 0
Proximal ε-globina
8 8 7 6 4 1
Proximal γ2-globina
4 4 2 2 2 1
Proximal γ1-globina
5 5 2 2 2 1
Proximal δ-globina
3 3 3 3 3 1
Proximal β-globina
2 2 2 2 2 1
3� HS1 1 1 0 0 1 0
De otra forma, las secuencias de regulación colectadas a través de TransFac® se
pueden agrupar según la ubicación de estas en el cluster de β-globina (Tabla 8).
Además, TransFac® señala para las secuencias caracterizadas en humano, cuales
hacen parte de los sitios hipersensitivos HS2, HS3 y HS4. Las coordenadas de estas
secuencias incluidas en un HS están dentro del rango de las coordenadas encontradas
para cada para sitio hipersensitivo accedido a través de la revisión bibliográfica
(exceptuando el trabajo de �High-throughput� de Dorschner, M. D. et al. 2004), por
tanto hay coincidencia entre la información de la revisión bibliográfica y la encontrada
en TransFac®.

82
Tabla 8. Rango de coordenadas de las regiones de regulación determinadas según los
sitios a donde se ubican las secuencias colectadas a partir de TransFac®. No se
incluye el pollo por no tener conservadas la mayoría de las secuencias de estas
regiones. Región Coordenadas
Humano Coordenadas Chimpancé
Coordenadas Ratón
Coordenadas Rata
Coordenadas Perro
Corriente abajo HBB
5202483-5202696
5028585-5028798
103685209-103685418
161616569-161616652
31296253-31296466
Exon1 HBB 5204380-5204608
5030482-5030710
103687257-103687485
161619720-161619948
31297817-31298045
Exon 2 HBB 5204734-5204880
5030836-5030982
103687597-103687745
161620053-161620201
31298169-31298315
Intron 1 HBB
5203536-5204375
5029638-5030477
103686609-103687252
161619043-161619715
31297226-31297812
Intron2 HBB
5204608-5204734
5030710-5030836
103687485-103687597
161619948-161620053
31298045-31298169
Corriente arriba HBB
5204880-5205425
5030982-5031548
103687745-103688293
161620201-161620656
31298315-31298790
Entre HBD-HBG
5221423-5221562
5049901-5050040
103717008-103717110
161636424-161636526
31312716-31312819
HS2 5258443-5258643
5088758-5088966
103734793-103734989
161658818-161659019
31330572-31330777
HS3 5262510-5262723
5092796-5093004
103739657-103739851
161662195-161662389
31334571-31334765
HS4 5265500-5265617
5095786-5095903
103744085-103744201
161667373-161667484
31337106-31337204
Por otro lado, de los sitios de regulación caracterizados en pollo y obtenidos a través
de TransFac® (Números 57 a 80 en Anexo 3) siete secuencias son descritas como
parte del sitio hipersensitivo HS2 (Figura 18) pero ninguno de estos sitios se han
conservado en mamíferos, al igual que ninguno de los sitios caracterizados como parte
de HS en mamíferos se encuentra conservado en pollo. Por este motivo no es posible
estimar la concordancia entre las dos fuentes de datos (TransFac® y revisión
bibliográfica) en cuanto a LCR en pollo. Solo se puede afirmar, a través de esta
información, que el LCR de pollo y mamíferos no se encuentra conservado en por lo
menos esas siete secuencias caracterizadas en pollo que hacen parte del HS2 y no
están presentes en mamíferos así como, en las secuencias que conforman HS2, HS3,
HS4 en humano y no están presentes en pollo.

83
6.4.1. Conservación del orden en que se ubican los sitios de regulación en cada
especie
Los sitios de unión a factores de transcripción generalmente están compuestos de 6 a
8pb (Hardison, R. C. 1998), en estos sitios tan cortos pueden ocurrir algunas
variaciones en su secuencia sin afectar la afinidad de la unión al factor de
transcripción. Así, un sitio de unión funcional puede estar formado de 6 nucleótidos
donde solo 4 coinciden en un alineamiento. Secuencias que cumplen este criterio
(alinear 4 de 6) ocurren al azar y con mucha frecuencia en toda la extensión de un
genoma haciendo difícil diferenciar sitios de unión funcionales de alineamientos al
azar, por esto, de un grupo de secuencias de regulación conservados en varias especies
es importante notar si el orden de estos sitios coincide, así como la distancia entre cada
sitio. En el anexo 7 se muestra el orden de los sitios de regulación conservados.
Al comparar el orden de los sitios de regulación (Anexo 7), se observa que en humano
y chimpancé la distribución de los sitios en el cluster es la mas conservada es decir
que estas dos especies comparten la mayoría de secuencias y estas mantienen en el
mismo orden, lo cual era de esperarse pues la similitud entre estos dos genomas es
muy alta. En cuanto a las secuencias conservadas entre humano, ratón y perro se
mantiene el mismo orden en la mayoría de estas. Sin embargo se observa
reorganización de los sitios de unión al factor de transcripción B1, caracterizados en
ratón, CACCC-binding factor y BP1, BP2, caracterizados en humano. De otra forma,
todos los sitios de unión a GATA-1 conservan el mismo orden en las tres especies e
incluso rata donde se presenta la mayor reorganización del orden de los sitios de
regulación lo cual puede deberse a que en esta especie el cluster ha tenido eventos de
duplicación y conversión (Okumura, S. et al. 2004; Satoh, H. et al. 1999) y se estima
que es un cluster heterogéneo respecto al mismo en las otras especies.
En cuanto a las cinco secuencias que siendo caracterizadas en humano y ratón están
conservadas en pollo hay reorganización de dos de estas en el pollo (Figura 19) y

84
además de la diferencia en orden, en humano y en ratón estos sitios están rodeados de
otras secuencias de regulación que en pollo no están conservadas.
Figura 19. Orden de tres sitios de regulación presentes en Humano, chimpancé, ratón,
rata y pollo, A muestra el orden y la posición de los sitios en el cluster de β-globina en
mamíferos. B muestra la posición y orden de las tres secuencias de regulación en
Pollo. Nótese que además de la diferencia en orden la distancia entre las secuencias es
mayor en Pollo.
De otra forma, en el cluster de β-globina en pollo se ha caracterizado un elemento de
regulación que se ubica entre los genes ε- globina y β-globina, denominado �enhancer�
β/ε, este elemento esta equidistante a los dos genes y se ha reportado competencia
entre los promotores de los dos genes para interactuar con el �enhancer� (Reitman, M.
et al. 1993; Hardison R.C. 1998). Se cree que la interacción de este elemento de
regulación con uno u otro promotor determina que gen es transcrito en una
determinada etapa del desarrollo (Reitman, M. et al. 1993; Recillas-Targa, F. et al.
2002). Dentro de las secuencias de regulación colectadas a partir de TransFac, 10
sitios de regulación se ubican entre los genes ε- globina y β-globina, a estos se unen
los factores de transcripción TFIID, GATA-1, TBP, PPUR, NFI/CTF, PYR, Sp1,
CACCC-binding factor y BGp1-G, debido a su ubicación en el cluster de pollo, estas
secuencias de regulación podrían estar relacionadas con el �enhancer� β/ε. De estos 10
sitios, 9 están presentes en humano, 8 en chimpancé, 7 en ratón y 6 en rata,
conservando en todos el mismo orden que en pollo, a excepción de una secuencia en
humano. Por otro lado, en rata, se mantiene el orden, pero entre uno y otro sitio
0 200 400 600 800 1000bp
GATA-1BP1,BP2 CACCC Bf
β-globin
GATA-1 BP1,BP2CACCC Bf
0 2000 4000 6000 8000 10000bp
ε-globin γ-globin
A
B

85
homologo a los del pollo, se ubican también otras secuencias de regulación homologas
a sitios caracterizados tanto en humano como en ratón pero en estas especies los sitios
no se ubican cerca a los caracterizados en pollo (Figura 20).
Figura 20. Conservación del orden de secuencias de regulación caracterizadas en
pollo (en pollo las secuencias están en la cadena positiva mientras que en mamíferos
están en la negativa, pero el orden 3� a 5� se mantiene). En humano se conservan 9 de
las cuales dos están en otra posición (sitio de unión a TFDII y NFI/CTF); en
chimpancé hay 8 secuencias conservadas, el sitio para TFIID que en humano estaba en
otra posición no esta conservado en chimpancé. En ratón hay 7 secuencias (se pierden
los sitios para TFDII y NFI/CTF) todas en el mismo orden respecto al pollo. En rata,
las secuencias que están conservadas con el pollo están en el mismo orden pero
rodeadas de otras secuencias caracterizadas en humano y ratón, secuencia que en
cuyas especias se ubican en otra región del cluster.
BP1, BP2
Sp1 GATA-1, TBP TFIID
PPYR CACCC-Binding factor
GATA-1 GATA-1
161,620,200 161,620,290 161,620,230
TFIID PPUR
Coordenadas en cromosoma 1 En Rata
199,436,650 199,436,750 199,436,850
Sp1
CACCC-Binding Factor
GATA-1, TBP TFIID
TFIID
NFI/CTF
PPYR
TFIIDPPURBGP1, G factor
Coordenadas en cromosoma 1 En Pollo
TFIID
5,221,400 5,221,450 5,221,500 5,421,550
CACCC- Binding factor
GATA-1, TBP TFIID
NFT/CTF
TFIID Sp1
PPUR
PPYR
Coordenadas en cromosoma 11 en Humano
5,049,900 5,049,950 5,050,000 5,050,050
TFIID
GATA-1, TBP TFIID
CACCC- Binding factor
NFT/CTF
Sp1
PPYR
TFIID
PPUR
Coordenadas en cromosoma 11 en Chimpancé
Coordenadas en cromosoma 7 En Ratón
103,717,100103,717,000
PPUR PPYR
Sp1
CACCC-Binding factor
GATA-1, TBP TFIID
TFDII
TFDII

86
Además de que la mayoría de secuencias, que en pollo podrían estar haciendo parte
del �enhancer� β/ε, están conservadas en humano, chimpancé, ratón y rata
manteniendo el mismo orden, también hay homología en cuanto a la región en donde
se ubica el conjunto de secuencias (exceptuando en rata) así, mientras que en pollo el
�enhancer� β/ε se ubica entre los genes β- y ε-globina, en humano y chimpancé las
secuencias se ubican entre los genes γ1- y δ- globina y en ratón entre γ- y ε- globina,
esto se puede representar que en mamíferos existe un �enhancer� homologo al
�enhancer� β/ε descrito en pollo. Es de notar que, en los dos grupos (aves y
mamíferos) la región conservada se encuentra entre dos genes uno de expresión
embrionaria y otro de expresión adulta.
Contrario a lo observado al ubicar las secuencias conservadas entre mamíferos y pollo,
Hardison, R. C. (1998) reporta que al realizar alineamientos de secuencias largas del
cluster de β-globina en pollo y en mamíferos (humano y ratón) no hay similitudes
significativas en las regiones de promotores y en las de elementos de control distal. El
encuentra que ninguna porción del cluster de β-globina en humano se alinea con las
secuencias del �enhancer� β/ε, así como tampoco hay alineamientos con las secuencias
del LCR. Sin embargo, Hardison reconoce la dificultad de ajustar el criterio de
alineamiento a las características de las secuencias de regulación. Por esto, es posible
que las comparaciones realizadas por Hardison, R. C. (1998) no hayan detectado
homología donde si existía. Por otro lado, en este mismo estudio de Hardison, R. C.
(1998) se propone que en mamíferos la región homologa a el �enhancer� β/ε se ubica
en HS2 por encontrar allí dos sitios de unión a GATA-1 y AP1/NFE2, sitios que
también se han descrito en el �enhancer� β/ε. Sin embargo, es necesario considerar que
la presencia de sitios de unión a dos factores de transcripción comunes no es
concluyente para determinar que dos regiones sean homologas especialmente cuando
secuencias de unión al factor de transcripción GATA-1 se encuentran varias veces en
toda la extensión del cluster.

87
Por lo anterior tiene mas soporte concluir que, de existir un sitio homologo en
mamíferos al �enhancer� β/ε de pollo, este se ubique entre dos genes del cluster y no
en un sitio hipersensitivo, y además que su homología no este restringida a los sitios
de unión sino también al orden de estos en el cluster. Así pues, según los resultados
obtenidos, se propone que, el �enhancer� β/ε caracterizado en pollo, tiene un posible
sitio homologo en los mamíferos en donde no solo se han conservado los sitios de
unión sino su orden y su ubicación en el cluster.
Estudios de Wheeler, D. y colaboradores (2001) donde los alineamientos de
secuencias del cluster de β-globina en pollo y mamíferos no muestran similitud
significativa aunque estos compartan sitios de unión a factores de transcripción
comunes, sugieren que los elementos de regulación del cluster de β-globina en
mamíferos y aves pueden compartir un ancestro común que ha divergido tanto que los
alineamientos de secuencias no alcanzan a detectar similitudes o que, por el contrario,
han aparecido independientemente en cada grupo implicando con esto la evolución
convergente de elementos de regulación en estos dos grupos.
6.4.2. Locus de la región de control
El locus de la región de control (LCR) se define operacionalmente por su capacidad de
inducir la expresión de la familia de genes de β-globina de manera especifica al tejido.
El LCR del cluster de β-globina en humano se localiza entre 6 a 22kb en el extremo
5� del primer gen del cluster (ε-globina) y consiste en cinco sitios hipersensitivos a
DNasa I (HS), los sitios HS 1 a 4 se forman únicamente en glóbulos rojos mientras
que HS5 se encuentra en varios linajes celulares (Li, Q. et al. 2002). En ausencia del
LCR la trascripción del gen humano β-globina es menos que el 1%. En experimentos
con ratones transgenicos donde incluyen en el cluster de β-globina sitios HS se ha
observado que se incrementa sustancialmente la expresión del gen beta, indicando que
LCR tiene una fuerte actividad de �enhancer� distal. Se ha descrito que dicha

88
actividad del LCR en el cluster de β-globina reside en HS2, HS3 y HS4, mientras que
HS5 presenta características de aislador (Molete, J. M. et al. 2002; Hardison, R. C. et
al. 1997; Bender, M. A. et al. 2006; Farrel, C. M. et al. 2002).
De la totalidad de sitios de regulación colectados algunos hacen parte de sitios
hipersensitivos (según anotación en TransFac® y revisión bibliográfica) con lo cual
fue posible ubicar tres de estos sitios en mamíferos y dos en pollo. Así, se encuentra
que las regiones descritas en TransFac® como parte de los sitios hipersensitivos HS2,
HS3 y HS4 en humano están conservadas en los mamíferos del estudio pero no en
pollo. En humano, chimpancé, ratón, rata, perro las regiones HS se ubican en el mismo
orden siendo la mas 5� HS4, seguida de HS3 y HS2, también se mantienen
conservadas en su orden las secuencias de regulación que hacen parte de cada sitio
hipersensitivo.
En pollo se mapeo el sitio hipersensitivo HS2 con base en las descripciones de
TransFac para siete secuencias de regulación, y el sitio HS4 con base en la
caracterización de Chung, J. H. et al. (1997); y Recillas-Targa, F. et al. (2002)
ninguno de estos sitios se encontró conservado en mamífero.
Molete, J. M. y colaboradores (2002) describen que el centro funcional de cada sitio
hipersensitivo es un fragmento de 100pb, definido como cantidad mínima DNA
necesaria para conferirle funcionalidad al HS, esto con base en experimentos
realizados en ratones transgenicos donde incluyen fragmentos de HS midiendo en cada
caso la expresión de los genes del cluster. El tamaño promedio de los sitios
reconocidos en este estudio como hipersensitivos es 250pb.
Comparaciones de las secuencias de el LCR en mamíferos (Hardison, R. C. et al.
1997; Molete, J. M. et al. 2002) muestran que la conservación de los sitios HS no esta
confinada a los centros de ~100pb sino que se hay amplias regiones homologas entre
los sitios hipersensitivos. Esto sugiere que las secuencias entre los HS son necesarias

89
para algún aspecto de la regulación ejercida por LCR y por eso dichos fragmentos de
DNA han sido conservados. En este orden de ideas Molete, J. M. y colaboradores
(2002) han propuesto que los centros HS interactúan en sinergia para incrementar la
expresión de los genes del cluster de β-globina y que para esta interacción son
necesarias las secuencias Inter.- HS. Molete, J. M. et al. (2002) sugiere que el DNA
entre los sitios hipersensitivos no ejerce función de regulación por si mismo, pero que
en este pueden encontrarse sitios de unión a factores de transcripción que son
necesarios para activar el funcionamiento de los HS, actuando todo el LCR como una
unidad integrada (�hollocomplex�).
Tabla 9. Sitios de unión a factores de transcripción reportados en la literatura y sitios
ubicados según las secuencias de regulación colectadas en este estudio.
Sitio hipersensitivo
Función Sitios de unión a factores de
trascripción
Sitios de unión ubicados según datos colectados
HS5
Aislador (Farrel et al, 2002)
CTCF, GATA-1, NF-E2 (Farrel et al, 2002;Li et al. 1999)
Ninguno
HS4
Activación de la transcripción (Molete et al. 2001)
GATA-1, AP1/NFE2 (Molete et al. 2001)
Cp2a
HS3
Activación de la transcripción conformación abierta de la cromatina
GATA-1, AP1/NFE2, YY1, EKLF (Molete et al. 2002; Shelton et al. 1997)
GATA-1, CAC-Binding protein, NF-E2
HS2
Actividad de �enhancer�
GATA-1, NF-E2, Sp1, AP-1, USF, EKLF (Harju et al. 2005)
GATA-1, AP-1,c-Fos, c-Jun, MafG, MafG:MafG, NF-E2, Nrf, Nrf1:MafG, Nrf2, Nrf2:MafG
HS1
No presenta actividad de activación de la transcripción
GATA-1 (Wheeler et al. 2005)
Ninguno

90
A pesar de la posible ubicación de secuencias de regulación en las regiones entre los
sitios hipersensitivos, ninguno de los sitios de regulación colectados en este estudio se
localiza en esas regiones del cluster. Además, muchos de los sitios de unión reportados
para cada sitio hipersensitivo no se lograron ubicar en este estudio. En la revisión de la
literatura hay caracterizaciones de sitios de unión en los HS pero no hay números de
accesión a secuencias y coordenadas lo cual imposibilita la ubicación de dichos sitios.
En la tabla 9 se estable comparación entre los sitios ubicados y los reportados en la
literatura por cada sitio hipersensitivo en el LCR de mamíferos.
6.4.3. Aisladores
Los aisladores son secuencias de DNA que protegen el locus de elementos de
regulación de genes vecinos la regulación inapropiada de los genes dentro del locus.
Uno de los aisladores mejor caracterizado en vertebrados es el HS4 del LCR del
cluster de β-globina en aves. Este aislador cumple las dos funciones típicas asignadas
a este tipo de elementos: es una barrera en la cromatina y bloquea la actividad de
�enhancers� externos.
Chung, J. H. y colaboradores (1997) caracterizaron la región HS4 de 1.2kb en pollo,
encontrando que la funcionalidad del aislador estaba restringida a un centro de 250pb
y mapearon dentro de estas 250pb cuatro �footprints� para determinar que secuencias
de regulación se ubicaban allí. De esta forma caracterizaron el sitio de unión al factor
de transcripción CTCF el cual es conocido por presentar actividad de activación y
represión de la transcripción.
En este estudio con base a los números de accesión a las secuencias de los �Footprints�
caracterizados por Chung, J. H. y colaboradores (1997) (No. accesión U78775, Anexo
cuatro secuencias de regulación 118 a 122), se ubico el sitio de unión al factor CTCF
en pollo y se hicieron alineamientos con esta secuencia para buscar el sitio homologo
en mamíferos. Los resultados de BLAT no mostraron algún alineamiento significativo

91
de esta secuencia (�Footprint� II) con los genomas de mamíferos. Es posible que el
cluster de β-globina en mamíferos y aves haya divergido tanto que el alineamiento no
reconozca el sitio homologo a HS4 de pollo en mamíferos.
Por otro lado, Farrel, C. M. y colaboradores (2002) caracterizaron el sitio de unión al
factor de transcripción CTCF en humano y ratón, encontrando que en mamíferos esta
secuencia de unión se ubica en el sitio hipersensitivo HS5. De manera similar
Tanimoto, K. et al. (2003) demostraron que HS5 en mamíferos presenta actividad de
aislador en el cluster de β-globina en mamíferos y que dicha actividad es dependiente
del sitio para el factor CTCF y es especifica al estado del desarrollo.
Tanto en los estudios de Farrel, C. M. et al (2002) y Tanimoto, K. y colaboradores
(2003) se considera que la región HS5 en mamíferos es ortóloga a la HS4 en aves. Sin
embargo, de las secuencias colectadas en este estudio, ninguno de los sitios ubicados
en HS5 en mamíferos esta conservado en pollo y, como se menciono antes, la
secuencia de HS4 de pollo no se alinea a ninguna región del cluster en mamíferos. Es
posible que en los dos grupos se hubiera adquirido un elemento aislante por
convergencia o que, por el contrario, las regiones si sean ortólogas pero que las
secuencias hayan divergido mucho. Teniendo en cuenta que CTCF es una proteína
altamente conservada y que se une a una amplia variedad de secuencias (Farrel, C. M.
et al. 2002), la divergencia de las secuencias de los aislantes a partir de una secuencia
común no impediría que se mantuviera la funcionalidad de la región, pues CTCF
adquirió la posibilidad de unirse a varias secuencias.
De todas las secuencias colectadas, es de notar que ninguna se ubica hacia el extremo
3� del cluster a pesar de que se han caracterizado aisladores en la región 3� del locus,
3�HS1 y 3�HS en mamíferos y en aves respectivamente. En mamíferos el aislador
3�HS1 no tiene actividad �enhancer� pero es responsable de atenuar la expresión de γ-
globina en humano. 3�HS1 también tiene sitio de unión a CTCF tanto en humano
como en ratón. Por otro lado, en pollo 3�HS y 5�HS4 coinciden con los limites

92
estructurales del cluster, es decir mas allá de los aisladores hay diferencia en el estado
de metilación o acetilación de la cromatina respecto a este dentro del locus. Contrario
a lo observado en aves, en mamíferos, el estado estructural de la cromatina no cambia
aun en regiones alejadas de los sitios 5�HS5 y 3�HS1, por lo que se ha sugerido la
posible existencia de secuencias de regulación en zonas mas allá de los aisladores o
incluso la presencia de aisladores ubicados mas corriente arriba y corriente abajo en
cada extremo del cluster (Farrel, C. M. et al. 2002; Molete, J. M. et al. 2001, Molete,
J. M et al. 2002; Bender, M. A. et al. 2006).
En general la única característica que comparten los dos grupos a este respecto es la
presencia del sitio de unión a CTCF (Farrel, C. M. et al. 2002), sin embargo, a partir
de esta similitud los aisladores HS5 de mamífero y HS4 en pollo han sido
considerados secuencias ortólogas (Farrel, C. M. et al. 2002; Molete, J. M. et al. 2002)
cuando posiblemente han sido adquiridas por convergencia, pues que son mas las
diferencias entre estas regiones de los dos clusters que las similitudes.
7. CONCLUSIONES
Teniendo en cuenta que este estudio es la primera aproximación a la comparación de
secuencias de regulación del cluster de β-globina en humano, chimpancé, ratón, rata,
perro y pollo, la información colectada aquí permite obtener una idea general de la
organización del cluster en estas especias logrando un acercamiento al posible patrón
de conservación de los mecanismos de regulación de este cluster de genes.
Se colecto información respecto a las secuencias de unión y su ubicación en el cluster
de β-globina en aves y mamíferos, de 24 factores de transcripción que intervienen en
la regulación de estos genes. Sin embargo, no se colectaron secuencias para algunos
factores que han sido identificados en la regulación de genes de β-globina y a pesar de
que para algunos de estos factores de transcripción se hayan caracterizado sus
secuencias de unión, aun hace falta información para poder determinar el sitio en el

93
cluster a donde se unen. Esto representa que de la gran cantidad de información
disponible respecto a regulación de este cluster, solo de un porcentaje se pueden
extraer secuencias y estimar coordenadas de sitios de regulación.
Respecto al patrón de conservación de los sitios de regulación colectados, estudios
previos indican que no hay alineamientos representativos entre las secuencias no
codificantes del cluster de β-globina en mamíferos y aves, grupos que divergieron
hace aproximadamente 250 millones de años (Hardison, R. C. 1998). Sin embargo, la
colección de datos de este trabajo muestra que si bien la mayoría de secuencias de
regulación no se encuentran conservadas entre los dos grupos, la regulación de este
cluster de genes en ambos grupos involucra elementos de regulación similares. Se
reconocen dos elementos comunes en la regulación de este cluster en aves y
mamíferos: la presencia de aisladores con sitio de unión al factor CTCF en ambos
extremos del cluster. Y la presencia en mamíferos de un posible sitio homologo al
�enhancer� β/ε de pollo.
Estos dos elementos comunes encontrados en pollo y mamíferos llevan a concluir que
es posible que entre estos dos grupos existan más similitudes en los elementos de
regulación, pero probablemente los criterios establecidos en la búsqueda mediante
comparación de secuencias no permita detectarlas. Es probable que si se comparan las
secuencias del cluster de los dos grupos sometiendo el alineamiento por regiones no
tan extensas, se reconozcan homologías antes no detectadas debido a que estas se
pueden encontrar restringidas a fragmentos cortos en el cluster, mientras que toda la
extensión del cluster, como un todo, haya divergido.
En cuanto a la organización de los genes y de los sitios de regulación en el cluster de
β-globina en los mamíferos, se puede concluir que se observa mayor variación en el
cluster de rata. En este cluster pudo haber duplicación del gen gamma, evento que ha
sido descrito en primates antropoides y que se ha relacionado con un cambio en el
patrón de expresión de los genes. Cambio que, según la revisión bibliográfica también

94
sucedió en el cluster de rata lo cual justificaría la posible duplicación. Además de la
presencia de dos genes gamma en el cluster de rata, también la distribución de los
sitios de regulación que se ubican entre los genes del cluster es diferente. A pesar de
que mantiene el orden de las secuencias, en rata la mayoría de estas se ubican
corriente arriba del gen beta, mientras que en los otros mamíferos esos sitios de
regulación se ubican entre los genes β-δ-γ
Los resultados muestran que entre perro y pollo no se comparte ninguna secuencia de
regulación, esto puede deberse a que el genoma del perro no se encuentra tan bien
anotado como el genoma de los otros mamíferos incluidos en este estudio, es posible
que en perro como en los otros mamíferos, estas secuencias de regulación si se
encuentren pero que estén pero que coincidan con polimorfismos o con �gaps� en la
secuencia y por tal motivo no hayan sido alienadas.
También se concluye que la colección de secuencias de regulación del cluster de β-
globina permitió identificar en todos los mamíferos incluidos en el estudio los sitios
HS4, HS3 y HS2 a partir de secuencias caracterizadas en humano, estableciendo que
el LCR se encuentra conservado en este grupo. Además, es posible que haya
secuencias de regulación en las regiones entre los sitios hipersensitivos, pues según
Farrel, C. M. et al. (2002) y Molete, J. M. et al. (2002), estas regiones han sido
conservadas y se cree que intervienen en la interacción de los sitios HS permitiendo
que estos actúen en sinergia.
En cuanto a los elementos aislantes, se reconoce que en mamíferos el sitio
hipersensitivo 5�HS5 es homologo al sitio HS4 en pollo el cual ha sido ampliamente
caracterizado como aislador. Sin embargo, HS5 no fue ubicado en mamíferos así
como tampoco el sitio de unión al factor CTCF el cual lo caracteriza como elemento
aislador.

95
Es posible que en mamíferos haya secuencias de regulación mas allá del sitio
hipersensitivo HS5, debido a que este no coincide con los limites estructurales de la
cromatina, lo cual si sucede en pollo en donde el elemento aislante HS4 coincide con
cambio en la conformación de la cromatina.
Finalmente, las secuencias colectadas en este trabajo permiten determinar dentro del
cluster de β-globina las regiones de DNA no codificante que se han conservado en los
mamíferos y pollo así como reconocer los factores de transcripción que se unen a esas
secuencias conservadas esta información será útil para futuros estudios.
8. RECOMENDACIONES
Las secuencias colectadas permiten identificar, en el cluster de β- globina, algunas
regiones de DNA no codificante que presentan similitud entre las diferentes especies,
esto representa el primer paso en el estudio de la comparación de los patrones de
regulación de la expresión de los genes de este cluster. Se propone que las secuencias
de estas regiones sean sometidas a análisis computacional para la búsqueda de más
motivos de regulación. Las secuencias de DNA reconocidos por los factores de
transcripción son generalmente cortos y degenerados, por lo que su identificación con
algoritmos computacionales ha representado un reto; al haber limitado las regiones en
donde se pueden encontrar con mas frecuencias estos motivos y habiendo determinado
los motivos de los factores de transcripción se facilitara la búsqueda de mas sitios de
regulación en el cluster de beta globina.
Se propone también determinar el motivo consenso a cada factor de transcripción
cuyas secuencias de unión se colectaron en este trabajo, con el fin de determinar el
motivo más común de unión a cada factor de transcripción y usarlos en la búsqueda
de más sitios de regulación en el cluster de β-globina.

96
Debido a que según la revisión bibliográfica, las comparaciones de secuencias en
mamíferos muestran que hay alineamientos significativos mucho mas allá de los
limites del cluster, y debido a que este se encuentra en un dominio de cromatina
abierto en mamíferos, se propone buscar motivos de regulación en fragmentos de
DNA mas allá del sitio hipersensitivo 5� HS5 y 3�HS1 puesto que es posible que en
estas regiones se encuentre un elemento otro elemento aislante, además de mas sitios
de regulación. Así mismo, se propone buscar por medio de algoritmos
computacionales secuencias de regulación en las regiones de DNA entre los sitios
hipersensitivos debido que estas regiones se han conservado y existe la posibilidad de
que en ellas haya motivos de regulación.
En general son pocas las similitudes encontradas entre aves y mamíferos. Sin
embargo, se propone realizar alineamientos de la región que corresponde al
�enhancer� β/ε en pollo contra las secuencias de las regiones donde el elemento
homologo a este se pueda ubicar en mamíferos. Así mismo, se propone realizar el
mismo procedimiento con las secuencias de los sitios hipersensitivos caracterizados en
pollo y en mamíferos.
9. REFERENCIAS
Asano, H., Li, X. S., Stamatoyannopoulos, G. 1999. FKLF, a novel Kruppel-like factor that activates human embryonic and fetal β-like globin genes. Mol Cell Biol. 19:3571-3579 Blackwood, E. M., Kadonaga, J. T. 1998. Going the Distances: A current View of Enhancer Action. Science 281:60-63 Bender, M. A., Byron, R., Ragoczy, T., Telling, A., Bulger, M., Groudine, M. 2006. Flanking HS-62.5 and 3�HS1, and regions upstream of the LCR, are not required for β-globin transcription. Blood. 108 ( 4):1395-1401 Bulger, M., Schubeler, D., Bender, M. A., Hamilton, J., Farrell, C. M., Hardison, R. C., Groudine, M. 2003. A complex chromatin "landscape" revealed by patterns of nuclease sensitivity and histone modification within the mouse -globin locus. Mol. Cell. Biol. 23:5234�5244.

97
Butler, J. E., Kadonaga, J. T. 2001. Enhancer-promoter specificity mediated by DPE or TATA core promoter motifs. Genes Dev.(15):2515�2519.
Calhoun, V. C., Stathopoulos, A., Levine, M. 2002, Promoter-proximal tethering elements regulate enhancer-promoter specificity in the Drosophila Antennapedia complex. Proc Natl Acad Sci USA. 99:9243�9247
Chan, J. Y., Han, X. L., Kan, Y. W. 1993. Cloning of Nrf1, an NF-E2-related transcription factor, by genetic selection in yeast. Proc Natl Acad Sci. USA. 90:11371-11375 Choi, O. R., Engel, J. D. 1988. Developmental regulation of β-globin gene switching. Cell. 55:17-26 Chung, J. H., Bell A. C., Felsenfeld G. 1997. Characterization of the chicken β-globin insulator. Proc. Natl. Acad. Sci USA 94:575-580 Dorfman, D. M., Wilson, D. B., Bruns, G. A., Orkin, S. H. 1992. Human transcription factor GATA-2. Evidence for regulation of preproendothelin-1 gene expression in endothelial cells. J. Biol Chem. 267:1279-1285 Dorschner, M. D., Hawrylcz, M., Humbert, R., Wallace, J. C., Shafer, A., Kawamoto, J., Mack, J., Hall, R., Goldy, J., Sabo, P. J., Kohli, A., Li, Q., McArthur, M., Stamatoyannopoulos, J. A. 2004. High-throughput localization of functional elements by quantitative chromatin profiling. Nature Methods 1(3): 219-225. Enver, T., Raich, N., Ebens, A. J., Papayannopoulou, T., Costantini, F., Stamatoyannopoulos, G. 1990. Developmental regulation of human fetal-to-adult globin gene switching in transgenic mice. Nature. 344(6264):309-313 Epner, E., Reik, A., Cimbora, D., Telling , A., Bender, M. A., Fiering, S., Enver, T., Martin, D. I., Kennedy, M., Keller, G., croudine, M. 1998. The β-globin LCR is not necessary for an open chromatin structure or developmentally regulated transcription of the native mouse β-globin locus. Mol Cell Biol. 2:447-455 Farrel, C. M., West, A. G., Felsenfeld, G. 2002. Conserved CTCF Insulator Elements Flank the Mouse and Human β-Globin Loci. Mol Cell Biol. 22(11):3820-3831 Filipe, A., Li, Q., Deveaux, S., Godin, I., Romeo, P. H., Stamatoyannopoulos, G., Mignotte, V. 1999. Regulation of embryonic/fetal globin genes by nuclear hormone receptors: a novel perspective on hemoglobin switching. EMBO J. 18:687-697

98
Forrester, W. C., Epner, A., Driscoll, M. C., Enver, T., Brice, M., Papayannopoulou, T., Groudine, M. 1990. A deletion of the human β-globin locus activation region causes a major alteration in chromatin structure and replication across the entire β-globin locus. Genes Dev. 4:1637-1649 Fraser, P., Hurst, J., Collis, P., and Grosveld, F. 1990. DNase I hypersensitive sites 1, 2 and 3 of the human β-globin dominant control region direct position-independent expression. Nucleic Acids Res. 18: 3503�3508 Gaszner, M., Felsenfeld, G. 2006. Insulators: exploiting transcriptional and epigenetic mechanisms. Nat. Rev. Genet. 7(9):703-13. Hardison, R. C., Miller, W., 1993. Use of long sequence alignments to study the evolution and regulation of mammalian globin gene clusters. Mol Biol Evol. 10(1):73-102 Hardison, R. C. 1998. Hemoglobins from Bacteria to Man: Evolution of Different Patterns of Gene Expression. Journal of Experimental Biology. 201: 1099-1117 Hardison, R. C. 2001. New views of evolution and regulation of vertebrate beta-like globin gene clusters from an orphaned gene in marsupials. Proc. Natl. Acad. Sci. USA. 98:1327-1329 Harju, S., McQueen, K. J., Peterson, K. R. 2002 Minireview: Chromatin Structure and Control of β-like Globin Gene Switching. Experimental Biology and Medicine 227(9):683-700.
Holland, R. A., Gooley, A. A. 1997. Characterization of the embryonic globin chains of the marsupial Tammar wallaby, Macropus eugenii. 2nd. Eur J Biochem. (248):864�871
Holland, R. A., Gooley, A. A., Hope, R. M. 1998. Embryonic globins of the marsupial the Tammar Wallaby (Macropus eugenii): bird like and mammal like. Clin Exp Pharmacol Physiol. (25):740�744. Jane, S. M., Ney, P. A., Vanin, E. F., Gumucio, D. L., Niehuis, A. W. 1992. Identification of a stage selector element in the human γ-globin gene promoter that fosters preferential interaction with the 5� HS2 enhancer when in competition with the β-promoter. EMBO J. 11:2961-2969 Jensen, L. J., Jensen, T. S., de Lichtenberg, U., Brunak, S., Bork, P. 2006. Co-evolution of transcriptional and post-translational cell-cycle regulation. Nature. 443(7111):594-597

99
Kent, W. J. 2002. BLAT--the BLAST-like alignment tool. Genome Res. 12(4):656-664. Kim, A., Dean, A. 2004. Developmental stage differences in Chromatin sub domains of β-globin locus. Proc. Natl. Acad. Sci USA 101(10):7028-7033 King, D. D., Taylor J., Elnitski, L., Chiaromonte, F., Miller, W., Hardison, R. C. 2005. Evaluation of regulatory potential and conservation scores for detecting cis-regulatory modules in aligned mammalian genome sequences. Genome Res. 15:1051-1060 Kuhn, R. M., Karolchik, D., Zweig, A. S., Trumbower, H., Thomas, D. J., Thakkapallayil, A., Sugnet, C. W., Stanke, M., Smith, K. E., Siepel, A., Rosenbloom, K. R., Rhead, B., Raney, B. J., Pohl, A., Pederson, J. S., Hsu, F., Hinrichs, A. S., Harte, R. A., Diekhans, M., Clawson, H., Bejerano, G., Barber, G. P., Baertsch, R., Haussler, D., Kent, W. J. .2006. The UCSC genome browser database: Update 2007. Nucleic Acids Res. 00(Data base issue):D1-D6 Lam, L. T., Bresnick, E. H. 1996. A Novel DNA-Binding protein, HS2NF5, interacts with a functionally important sequence of the human β-globin locus control region. J. Biol. Chem. 271:32421-32429 Lemon, B. and Tjian, R. 2000. Orchestrated response: A symphony of transcription factors for gene control. Genes y Dev. (14): 2551-2569. Levine, M., Tjian, R., 2003. Transcription regulation and animal diversity. Nature. 424(6945):147-51 Li, J., Noguchi, C., Miller, W., Hardison, R. C., Schechter, A. 1998. Multiple regulatory elements in the 5'-flanking sequence of the human -globin gene. J. Biol. Chem. 273:10202�10209 Li, Q., Zhang, M., Han, H., Rohde, A., Stamatoyannopoulos, G. 2002. Evidence that DNase I hypersensitive site 5 of the human beta-globin locus control region functions as a chromosomal insulator in transgenic mice. Nucleic Acids Res. 30:2484-2491 Li, Q., Peterson, K. R., Fang, X., Stamatoyannopoulos, G. 2002.Locus Control Regions. Blood, (100): 3077-3086 Mariño-Ramírez, L., Jordan, I. K., Landsman, D. 2006. Multiple independent evolutionary solutions to core histone gene regulation. Genome Biol. 7(12):R122 Mariño-Ramírez, L., Kann, M. G., Shoemaker, B. A., Landsman, D. 2005. Histone structure and nucleosome stability. Expert Rev. Proteomics (5):719-29

100
Mariño-Ramírez, L., Spouge, J. L., Kanga, G. C., Landsman, D. 2004. Statistical analysis of over-represented words in human promoter sequences. Nucleic Acids Res. 32(3):949-958 Matys, V., Kel-Margoulis, O. V., Fricke, E., Liebich, I., Land, S., Barre-Dirrie, A., Reuter, I., Chekmenev, D., Krull, M., Hornischer, K., Voss, N., Stegmaier, P., Lewicki-Potapov, B., Saxel, H., Kel, A. E., Wingender, E. 2006. TRANSFAC® and its module TRANSCompel®: Transcriptional gene regulation in eukaryotes. Nucleic Acids Res. 34 (Data base issue): D108-D110 McClean, P. 1997. Control of Gene Expression In Eukaryotas [En linea]: Plcs: Intermediate Genetics.http://www.ndsu.nodak.edu/instruct/mcclean/ [Consultada 30 de septiembre de 2006] Merika, M., Orkin, S. H. 1993. DNA Binding specificity of GATA family transcription factors. Mol Cell Biol. 13:3999-4010 Molete, J. M., Petrykowska, H., Bouhassira, E. E., Feng, Y., Miller, W., Hardison, R. C., 2001. Sequences Flanking Hypersensitive Sites of the β globin Locus Control Region are required for Synergistic Enhancement. Mol Cell Biol. 21(9):2969-2980 Molete, J. M., Petrykowska, H., Sigg, M., Miller, W., Hardison, R. C. 2002. Functional and binding studies of HS3.2 of the beta-globin locus control region. Gene (283): 185-197 Ney, P. A., Sorrentino, B. P., Mc Donagh, K. T., Nienhuis, A. W. 1990. Tandem AP-1-Binding sites within the human β-globin dominant control region functions as an inducible enhancer in erythroid cells. Gene Dev. 4:993-1006 Okumura S., Fujii, H., Inokuchi, N., Watanabe, M., Nishino, T., Okasaki, T. 2004. Molecular cloning and characterization of three adult rat β-globin gene promoters. Biochimica et biophysica Acta (1678):145-149 Ouyang, L., Chen, X., Bieker, J. J. 1998. Regulation of Erythroid Kruppel-like factor (EKLF) transcriptional activity by phosphorilation of a protein kinase II site within its interaction domain. J. Biol. Chem. 273:23019-23025 Raich, N., Clegg, C. H., Grofti, J., Romeo, P. H., Stamatoyannopoulos, G. 1995. GATA-1 and YY1 are developmental repressors of the human ε-globin gene. EMBO J. 14:801-809 Recillas-Targa, F., Pikaart, M. J., Burgess-Beusse, B., Bell, A. C., Litt, M. D, West, A. G., Gaszner, M., Felsenfeld, G. 2002. Position-effect protection and enhancer blocking

101
by the chicken beta-globin insulator are separable activities. Proc Natl Acad Sci U S A. 99(10):6883-6888. Reitman, M., Grasso, J. A., Blumentahl, R., Lewit, P. 1993. Primary sequence, evolution, and repetitive elements of the Gallus gallus (chicken) beta-globin cluster. Genomics. 18:616�626. Ryan, T. M., Sun, C. W., Ren, J., Townes, T. M. 2000. Human gamma-globin gene promoter element regulates human beta-globin gene developmental specificity. Nucleic Acids Res. 28(14):2736-2740. Satoh, H., Inokuchi, N., Nagae, Y., Okasaki, T. 1999. Organization, structure and evolution of the Nonadult rat β-globin gene cluster. J. Mol. Evol. 49:122-129 Stamatoyannopoulos, G., Josephson, B., Zhang, J. U., and Li, Q. 1993. Developmental regulation of human -globin gene in transgenic mice. Mol. Cell. Biol. 13:7636�7644 Struhl, K. 2001. Perspectives: Gene Regulation. A paradigm for Precision. Science 293: 1054-1055 Talbot, D., Philpsen, S., Fraser, P., Grosveld, F. 1990 Detailed Analysis of the site 3 region of the human β-globin dominant control region. EMBO J. 9: 2169-2177 Tanay, A., Regev, A., Shamir, R. 2005. Conservation and evolvability in regulatory networks: the evolution of ribosomal regulation in yeast. Proc Natl Acad Sci. USA. 102(20):7203-7208 Tang, D. C., Ebb, D., Hardison, R. C., and Rodgers, G. P. 1997. Restoration of the CCAAT box and insertion of the CACCC motif activate d-globin gene expression. Blood 90:421�427. Tanimoto, K., Sugiera, A., Omori, A., Felsenfeld, G., Ángel, J. D., Fukamizu, A. 2003. Human β- globin Locus Control region HS5 Contains CTCF- and Developmental Stage-Dependent Enhancer-Blocking Activity in Erythroid Cells. Mol. Cell Biol. 23(24):8946- 8952 Tanimoto K., Liu, Q., Bungert, J. and Engel, J. D. 1999. Effects of altered gene order or orientation of the locus control region on human beta-globin gene expression in mice. Nature. (398):344�348 Tanimoto, K., Liu, Q., Grosveld, F., Bungert, J., Engel, J. D. 2000. Context-dependent EKLF responsiveness defines the developmental specificity of the human ε-globin gene in erythroid cells of YAC transgenic mice. Gene Dev. 14:2778-2794

102
Tsang, A. P., Visvader, J. E., Turner, C. A., Fujiwara, Y., Yu, C., Weiss, M. J., Crossley, M., Orkin, S.H. 1997. FOG, a multiple zinc finger protein , acts as cofactor for transcription factor GATA-1 in erythroid and megakaryocytic differentiation. Cell. 90:109-119
Welch, J., Waltts, J. A., Vakoc, C. R., Yao, Y., Wang, H., Hardison, R. C., Blobel, G. A., Chodosh, L. A., Weiss, M. J. 2004. Global regulation of Erythroid gene expression by transcription factor GATA-1. BLOOD. 104(10):3136-3147
West, A. G., Gaszner, M., Felsenfeld, G. (2002) Insulators: Many functions, many mechanisms. Genes Dev. (16):271�288 Wheeler, D., Hope, R., Cooper, S. B., Dolman, G., Webb, G. C., Bottema, C. D., Gooley, A. A., Goodman, M., Holland R. A.. 2001. An orphaned mammalian beta-globin gene of ancient evolutionary origin. Proc Natl Acad Sci USA. 98:1101-1106 Wijgerde, M., Gribnau, J., Nuez, B., Philpsen, S., Grosveld, F., Fraser, P. 1996. The role of EKLF in Human β-globin gene competition. Genes Dev. 10:2894-2902 Wray, G. A., Hahn, M. W., Abouheif, E., Balhoff, J. P., Pizer, M., Rockman, M. V., Romano, L.A. 2003. The Evolution of Transcriptional Regulation in Eukaryotes. Mol Biol Evol. 20(9):1377-1419. Wright, S., Rosenthal, A., Flavell, R., Grosveld, F. 1984. DNA sequences required for regulated expression of -globin genes in murine erythroleukemia cells. Cell. 38:265�273 Yu, C. Y., Motamed, K., Chen, J., Bailey, A.D., Shen, C. K. J. 1991. The CACC box upstream of human embryonic globin gene binds Sp1 and is a functional promoter element in vitro and in vivo. J. Biol. Chem. 266:8907�8915 Zhou, W., Clouston, D. R., Wang, X., Cerruti, L., Cunningham, J. M., Jane, S. M. 2000. Induction of Human fetal globin gene expression by a novel erythroid factor, NF-E4. Mol Cell Biol. 20:7662-7672